AUTOMATISATIE VAN HET DISK DIFFUSIE ANTIBIOGRAM
MET BEHULP VAN DE WASP®
Lisa De Meerleer
Masterproef in de opleiding Master in de Farmaceutische Zorg
AUTOMATISATIE VAN HET DISK DIFFUSIE ANTIBIOGRAM
MET BEHULP VAN DE WASP®
Lisa De Meerleer
Masterproef in de opleiding Master in de Farmaceutische Zorg
AUTEURSRECHT
“De auteur en de promotor geven de toelating deze masterproef voor consultatie beschikbaar te stellen en delen ervan te kopiëren voor persoonlijk gebruik. Elk ander gebruik valt onder de beperkingen van het auteursrecht, in het bijzonder met betrekking tot de verplichting uitdrukkelijk de bron te vermelden bij het aanhalen van de resultaten uit deze masterproef.”
Mei 2020
Promotor Auteur
PREAMBULE
Deze preambule werd in overleg tussen de student en de promotor opgesteld en door beiden goedgekeurd. De planning voor de automatisatie van het disk diffusie antibiogram met behulp van de WASP® verloopt als volgt. Eerst wordt het meest geschikte patroon en entoog van de entautomaat WASP® voor de automatische enting van disk diffusie antibiogrammen bepaald. Vervolgens worden WASP® geënte antibiogrammen vergeleken met antibiogrammen ingezet via de conventionele, manuele methode. Het onderzoek vergelijkt de benodigde tijsduur en resultaten tussen de twee methodes. Tot slot volgt een interpersoneelsvergelijking waarbij Medisch Laboratoriumtechnologen met behulp van AdagioTM de zone diameters van antibiogrammen aflezen. Tevens worden ook testen ingezet door middel van de WASP®: WASP® geënte platen met manueel aangebrachte E-test strips worden vergeleken met de manuele methode.
Vanaf 16 maart 2020 werd het experimenteel werk ten gevolge van de coronamaatregelen stopgezet. Het grootste deel van het onderzoek werd evenwel reeds uitgevoerd. Voor de disk diffusie methodevergelijking zouden normaal van elke species tien stalen getest moeten zijn en ook E-testen ingezet d.m.v. de WASP® werden niet uitgevoerd. Het onderzoek werd afgewerkt op basis van de reeds bekomen resultaten met een aanvullende literatuurstudie over genotypische gevoeligheidsbepalingen.
SAMENVATTING
Achtergrond: Antimicrobiële resistentie is een toenemend probleem waardoor de vraag naar het testen van de gevoeligheid van een kiem voor antibiotica steeds belangrijker wordt. De disk diffusie methode is een vaak toegepaste antimicrobiële gevoeligheidstest in de routinepraktijk van een Laboratorium voor Medische Microbiologie. Dagelijks besteden Medisch Laboratoriumtechnologen veel tijd aan het inzetten en aflezen van antibiogrammen, uitgevoerd met deze methode. Automatisatie van gevoeligheidstesten kan hiervoor een oplossing bieden door een tijdswinst te impliceren. De disk diffusie methode is momenteel semi-geautomatiseerd in het microbiologie lab van het UZ Gent aangezien na manuele handelingen de diameters van de inhibitiezones worden afgelezen met het semi-geautomatiseerd systeem AdagioTM.
Doelstelling: Er wordt nagegaan of de entautomaat WASP® een nuttige bijdrage kan leveren aan de verdere automatisatie van het disk diffusie antibiogram. De WASP® beschikt hiervoor over een disk carrousel, maar deze is niet geïnstalleerd op de huidig beschikbare configuratie in het UZ Gent. Daarom wordt in dit thesisonderzoek de WASP® enkel gebruikt voor de automatische enting van platen voor de disk diffusie, gevolgd door het manueel aanbrengen van antibioticumschijfjes en het plaatsen van de antibiogrammen in de incubator. Dit ter voorbereiding op verkennend onderzoek naar een eventuele latere implementatie van de nodige configuratie. Methoden: Eerst wordt bepaald welk entpatroon en welk entoog de beste resultaten geven voor enting door de WASP®. Zodra het juiste protocol op de WASP® is geïnstalleerd, kunnen antibiogrammen van diverse species worden ingezet, zowel via de manuele methode als de WASP® methode. De benodigde tijdsduur en zone diameters met bijhorende interpretatie worden tussen de twee methodes vergeleken. Tot slot volgt een interpersoneelsvergelijking waarbij zes antibiogrammen door iedereen van het lab worden afgelezen.
Resultaten: Gedurende het experiment worden antibiogrammen geënt door het 30 μL entoog in drie richtingen. Het wordt al snel duidelijk dat enting door de WASP® aanzienlijk meer tijd vergt dan de manuele methode. Ook andere aspecten van de door WASP® geënte antibiogrammen zorgen voor moeilijkheden: platen vertonen geen confluente bacteriegroei en de strepen van het entoog leiden tot verwarring bij aflezing van de zone diameters. In het algemeen zijn de diameters van de WASP® antibiogrammen kleiner dan de diameters van de manuele methode, wat ervoor zorgt dat een kiem sneller als resistent wordt gecategoriseerd.
Conclusie: Enkel antibiogrammen voor de disk diffusie methode automatisch laten enten door de WASP® blijkt geen voordelen te hebben. Het duurt lang, wat niet ideaal is voor gebruik in routine, en er treden moeilijkheden en fouten op bij aflezing van de zone diameters. Maximale percentages van discrepanties, om te besluiten of de twee methodes vergelijkbare resultaten opleveren, kunnen niet worden berekend wegens onvoldoende data. Het zou kunnen dat vanaf een bepaalde hoeveelheid stalen een geautomatiseerde enting wel nut heeft, maar daarvoor is verder onderzoek nodig.
DANKWOORD Ondanks dat mijn aanwezigheid in het Laboratorium voor Medische Microbiologie korter dan verwacht was, ben ik zeer blij dat ik deze mogelijkheid heb gekregen. Op korte tijd heb ik veel bijgeleerd en was het interessant om theorie eens toegepast in de dagdagelijkse routine te zien. Graag zou ik een aantal mensen willen bedanken die hebben geholpen aan het tot stand komen van deze masterproef. In de eerste plaats wil ik Prof. Dr. Stove bedanken voor zijn betrokkenheid, zinvolle opmerkingen en suggesties
gedurende het semester. In het bijzonder zou ik graag Prof. Dr. Boelens willen bedanken voor het meermaals nalezen van mijn masterproef en nuttige feedback te geven. Het was frappant om iemand die zo geboeid en enthousiast is voor het domein microbiologie te mogen ontmoeten. Oprecht dankuwel om in deze drukkere tijden tijd voor mij vrij te maken. Ook de laboranten van het UZ Gent zou ik graag bedanken. De eerste weken mocht ik meevolgen in de routine
en werd ik er ook bij betrokken. Ik kon altijd bij iedereen terecht voor uitleg en vragen. Tot slot wil ik mijn mama bedanken om mijn masterproef samen met mij na te lezen en mij veel tips te geven op vlak van schrijven.
INHOUDSOPGAVE
1. INLEIDING ... 1
1.1 SITUERING ... 1
1.2 ANTIMICROBIËLE GEVOELIGHEIDSTESTEN ... 3
1.2.1 Kiemen categoriseren: gevoelig, intermediair of resistent ... 3
1.2.2 Begrippen: MIC, klinisch breekpunt en ECOFF ... 4
1.2.3 Standaardiseren van antimicrobiële gevoeligheidstesten ... 5
1.2.4 Fenotypische gevoeligheidstesten ... 6
1.2.4.1 Broth dilutie methode ... 6
1.2.4.2 Gradiënt diffusie test ... 6
1.2.4.3 Disk diffusie methode ... 7
1.2.4.4 MALDI-TOF MS ... 8
1.2.5 Gentoypische gevoeligheidsbepalingen ... 11
1.2.5.1 PCR en real-time PCR ... 11
1.2.5.2 DNA-hybridisatie en DNA-microarray ... 13
1.2.5.3 Whole Genome Sequencing ... 14
1.2.6 Fenotypische versus genotypische gevoeligheidstesten ... 15
1.3 AUTOMATISATIE IN HET LABO MICROBIOLOGIE ... 16
1.3.1 Nood aan automatisatie ... 16
1.3.2 Huidige automatisatie ... 17
1.3.2.1 Liquid based microbiology ... 17
1.3.2.2 Identificatie met MALDI-TOF MS ... 18
1.3.3 Total Laboratory Automation ... 19
1.4 ENTEN MET DE WASP® in UZ Gent ... 19
1.5 AUTOMATISATIE ANTIMICROBIËLE GEVOELIGHEIDSTESTEN ... 21
1.5.1 Systemen voor de disk diffusie methode ... 21
1.5.2 Systemen voor de broth microdilutie ... 22
1.5.3 Geautomatiseerde broth-gebaseerde gevoeligheidstesten ... 22
1.5.3.1 Historiek ... 22
1.5.3.2 Vitek® en Vitek® 2 systeem ... 23
1.5.3.3 SensititreTM ARIS ... 24
1.5.3.4 MicroScan WalkAway® systeem ... 24
1.5.3.5 BD PhoenixTM ... 25
3. MATERIALEN EN METHODEN ... 27 3.1 PATROON EN ENTOOG ... 27 3.1.1 Materialen ... 27 3.1.2 Methoden ... 27 3.1.2.1 Dag 1 ... 28 3.1.2.2 Dag 2 ... 30
3.2 ANTIBIOGRAMMEN WASP® VERSUS MANUEEL ... 30
3.2.1 Materialen ... 30 3.2.2 Methoden ... 32 3.2.2.1 Dag 1 ... 32 3.2.2.2 Dag 2 ... 32 3.3 INTERPERSONEELSVERGELIJKING ... 32 3.3.1 Materialen ... 32 3.3.2 Methoden ... 33 3.3.2.1 Dag 1 ... 34 3.3.2.2 Dag 2 ... 34 3.4 BEGRIP DISCREPANTIE ... 34 3.4.1 Minor error ... 34 3.4.2 Major error ... 34
3.4.3 Very major error ... 35
4. RESULTATEN ... 36
4.1 PATROON EN ENTOOG ... 36
4.2 ANTIBIOGRAMMEN WASP® VERSUS MANUEEL ... 36
4.2.1 Snelheid ... 36 4.2.2 Zone diameters ... 38 4.3 INTERPERSONEELSVERGELIJKING ... 41 5. DISCUSSIE ... 46 6. CONCLUSIES ... 49 7. LITERATUURLIJST ... 50 BIJLAGEN ... I
LIJST MET GEBRUIKTE AFKORTINGEN AST: Antimicrobial Susceptibility Testing ARIS: Automatic Reading and Incubation System cDNA: complementair DNA
CLSI: Clinical Laboratory Standards Institute Cq: kwantificatiecyclus
CV: variatiecoëfficiënt d.m.v.: door middel van
ECOFF: Epidemiologische Cut-Off E-test: Epsilometer test
EUCAST: European Committee on Antimicrobial Susceptibility Testing I: intermediair
IU: International Unit
LIS: Laboratorium Informatica Systeem
MALDI-TOF MS: Matrix Assisted Laser Desorption Ionization- Time Of Flight Massaspectrometrie MBT-ASTRA®: MALDI Biotyper-Antibiotic Susceptibility Test Rapid Assay
MBT-RESIST®: MALDI Biotyper-Resistance Test with Stable Isotopes
MBT STAR®-BL: MALDI BiotyperSelective Testing Antimicrobial Resistance-Beta-Lactamase mE: minor error
ME: major error MH: Mueller-Hinton
MH-F: Mueller-Hinton Fastidious (MH gesupplementeerd met 5% gedefibrineerd paardenbloed en 20 mg/L β-NAD)
MIC: Minimaal Inhiberende Concentratie MLT: Medisch Laboratoriumtechnoloog
MRSA: Methicilline-Resistente Staphylococcus aureus m/z: massa/ladingsverhouding
NAD: Nicotinamide Adenine Dinucleotide NGS: Next-Generation Sequencing PBP: penicilline bindend proteïne PCR: Polymerase Chain Reaction R: resistent
RFU: Relative Fluorescence Units S: gevoelig
SD: standaarddeviatie
SensititreTM ARIS: Sensititre Automatic Reading and Incubation System TLA: Total Laboratory Automation
t.o.v.: ten opzichte van vME: very major error
WASP®: Walk Away Specimen Processor
1. INLEIDING 1.1 SITUERING
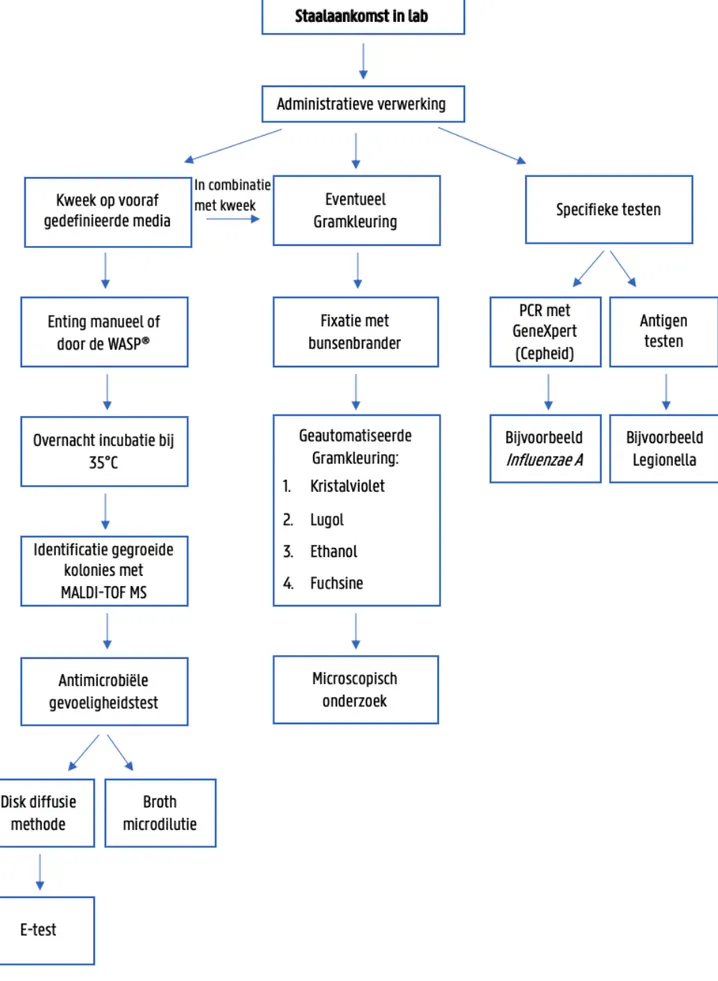
Het Laboratorium voor Medische Microbiologie speelt een centrale rol in de diagnose van bacteriële infecties en bij de optimalisatie van de antibiotische behandeling (1). In het lab van het UZ Gent worden dagelijks verschillende soorten stalen zoals urines, faeces, sputa en hemoculturen geanalyseerd op de aanwezigheid van ziekteverwerkers. Niet enkel bacteriën, maar ook schimmels, gisten en parasieten kunnen opgespoord worden. Stalen worden grotendeels onderzocht volgens dezelfde stappen (Figuur 1.1). Eerst wordt het staal, na aanmelding en administratieve verwerking, manueel of met de Walk Away Specimen Processor, (WASP®, Copan, Italië), geënt op vooraf gedefinieerde media. Voor elk staal worden telkens één of meerdere relevante media ingezet. Meestal wordt geopteerd voor een tryptic soy agar met 5% schapenbloed, kortweg een bloedplaat waarop diverse micro-organismen kunnen groeien, en eventueel een aantal selectieve, aanrijkings- en differentiële media. In sommige gevallen wordt voor microscopisch onderzoek een Gramkleuring uitgevoerd. De Gramkleuring is een differentiële kleuring die onderscheid maakt tussen Gram positieve en Gram negatieve bacteriën op basis van de structuur van de celwand (respectievelijk donkerpaars en rood aankleurend). Na overnacht incubatie van de geënte voedingsbodems bij 35°C analyseert de MLT (Medisch Laboratoriumtechnoloog) de groei op de media en worden verdachte kolonies verder uitgewerkt. Voor elk type staal ligt in procedures vast hoe de resultaten geïnterpreteerd moeten worden. De identificatie van de bacteriën gebeurt d.m.v. Matrix Assisted Laser Desorption Ionization - Time Of Flight Massaspectrometrie (MALDI-TOF MS) (2). Na identificatie van pathogene bacteriën kan het nuttig zijn om een antimicrobiële gevoeligheidsbepaling uit te voeren om voor een patiënt een gerichte antibiotische behandeling te kunnen garanderen. In het lab van UZ Gent is disk diffusie hiervoor de meest gebruikte methode en soms wordt voor een bepaalde bacterie-antibioticum-combinatie bijkomend een E-test ingezet. Een andere methode voor gevoeligheidsbepaling is de broth microdilutie. Deze wordt in het UZ Gent enkel gebruikt voor fungi en het testen van enkele welbepaalde antibiotica, zoals colistine (3).
1.2 ANTIMICROBIËLE GEVOELIGHEIDSTESTEN
1.2.1 Kiemen categoriseren: gevoelig, intermediair of resistent
Vooral in ziekenhuizen, waar veel resistente kiemen voorkomen, is het noodzakelijk om de gevoeligheid van een pathogeen t.o.v. antibiotica te bepalen. Een snel resultaat is belangrijk om zo vlug mogelijk een gerichte antibiotische behandeling te voorzien of de reeds gestarte behandeling te kunnen optimaliseren. Antimicrobiële gevoeligheidstesten of AST (Antimicrobial Susceptibility Testing) kunnen gebaseerd zijn op het fenotype of genotype van bacteriën. Momenteel zijn de fenotypische gevoeligheidstesten de gouden standaard (3). Broth microdilutie en disk diffusie zijn de meest gekende en gebruikte fenotypische methodes. Het doel van deze testen is te bepalen tot welke categorie een kiem behoort: gevoelig (S), intermediair (I) of resistent (R) voor welbepaalde antibiotica. De broth microdilutie geeft meer informatie door het rapporteren van een minimaal inhiberende concentratie (Paragraaf 1.2.2). Wanneer een kiem gecategoriseerd wordt als gevoelig voor een antibioticum betekent dit dat de kans op therapeutisch succes bij behandeling van een infectie met die kiem en het geteste antibioticum hoog is. De kans op therapeutisch falen van een behandeling is dan weer hoger bij een resistente kiem. De intermediaire categorie kan gezien worden als een grijze zone tussen S en R om ernstige categoriseerfouten te vermijden. Een intermediaire kiem vertoont minder respons tegenover een antibioticum dan de gevoelige categorie, maar behandeling ervan kan wel succesvol zijn bij het intensifiëren van de dosis of als het antibioticum geconcentreerd voorkomt ter hoogte van de plaats van infectie. Niet voor elke antibioticum-kiem-combinatie is een intermediaire zone gedefinieerd (4).
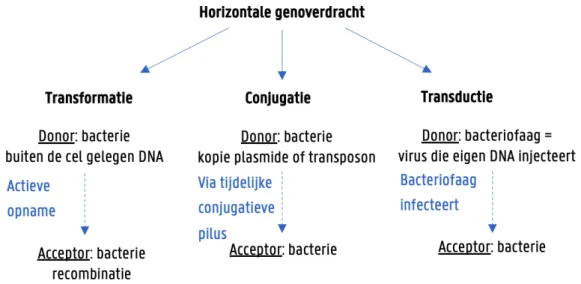
Resistentie kan zowel eigen zijn aan het species (intrinsieke resistentie) als verworven (ten gevolge van mutaties en antibioticumdruk). Voornamelijk de verworven resistentie maakt het steeds moeilijker om patiënten te behandelen met de meest gangbare antibiotica. Intrinsieke resistentie betekent dat een bacterie van nature ongevoelig is voor een antibioticum door een stabiele genetische eigenschap gecodeerd in het bacteriële DNA. Een bacterie kan resistentie ontwikkelen door mutaties of door het verwerven van nieuw genetisch materiaal. Genetische informatie kan op mobiele genetische elementen, zoals plasmiden en transposons, voorkomen en via een horizontale genoverdracht uitgewisseld worden binnen één soort of tussen verschillende species. Horizontale genoverdracht kan via drie mechanismen plaatsvinden: transformatie, conjugatie of transductie (Figuur 1.2) (5,6).
Er zijn verschillende bacteriële resistentiemechanismen (6): • Wijziging van het antimicrobieel doelwit in bacteriën • Enzymproductie waardoor inactivatie van antibiotica • Verhoogde expressie van efflux pompen
• Verandering in permeabiliteit van het celmembraam
Hoe meer een patiënt blootgesteld wordt aan antibiotica, hoe meer kans op het ontwikkelen van resistentie. Resistentie voorkomen, kan alleen door zo beperkt mogelijk antibiotica voor te schrijven en de richtlijnen met betrekking tot antibioticagebruik en infectiepreventie op te volgen. Zo rapporteert het lab bij welbepaalde patiëntengroepen en infectietypes niet steeds alle antibiotica waaraan een bacterie gevoelig is, maar enkel de initieel meest aanbevolen antibiotica. Dit om overgebruik van breedspectrum antibiotica en zo snellere evolutie naar resistentie te beperken. In geval van resistentie, allergie… moeten uiteraard wel andere antibiotica worden gerapporteerd (7).
Figuur 1.2: Horizontale genoverdracht (eigen illustratie).
1.2.2 Begrippen: MIC, klinisch breekpunt en ECOFF
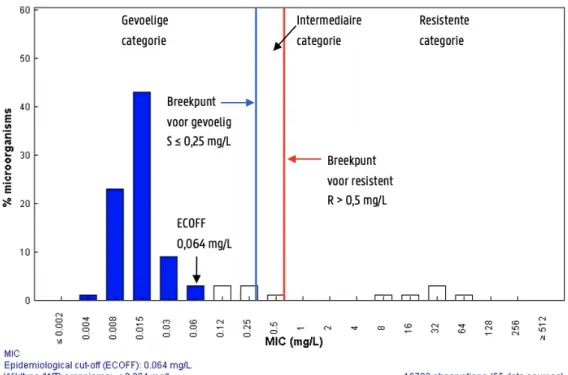
De meest gebruikte parameter bij de evaluatie van antimicrobiële gevoeligheid is de MIC (Minimaal Inhiberende Concentratie). De MIC, uitgedrukt in mg/L of μg/mL, wordt gedefinieerd als de laagste concentratie van een antibioticum dat de groei van de onderzochte bacterie inhibeert (5). Een MIC weerspiegelt niet noodzakelijk de reële werkzaamheid van een antibioticum. Dit hangt immers af van de gerelateerde farmocokinetische en farmacodynamische parameters. De farmacokinetiek beschrijft “wat het lichaam van de patiënt doet met het geneesmiddel” (absorptie, distributie, metabolisatie en eliminatie). Farmacodynamiek bestudeert “het effect van het geneesmiddel op het micro-organisme” (3,8). Klinisch breekpunten worden gebruikt om bacteriespecies d.m.v. MIC-waarden te categoriseren als gevoelig, intermediair of resistent voor een antibioticum. Deze in vitro bepaling heeft als doel om de doeltreffendheid van het antibioticum in vivo te voorspellen. Een MIC lager dan of gelijk aan de waarde van het breekpunt voor gevoelig, betekent dat de kiem gecategoriseerd kan worden als gevoelig. Een waarde hoger dan het klinisch breekpunt voor resistent
categoriseert een kiem als resistent. MIC’s tussen deze twee breekpunten vertegenwoordigen de intermediaire categorie (Figuur 1.3). Indien geen intermediaire categorie wordt gedefinieerd, is er slechts één klinisch breekpunt. Om verworven resistentiemechanismen op te sporen kan de ECOFF of Epidemiologische Cut-Off waarde worden bepaald. De ECOFF verwijst naar de MIC-waarde die de bovengrens van de populatie zonder verworven resistentiemechanismen (“wild type” populatie) aanduidt (Figuur 1.3). Dit betekent niet automatisch dat elke stam buiten de wild type populatie resistent is, want dat hangt af van het klinisch breekpunt. Een stam met een MIC hoger dan de ECOFF, maar lager dan het klinisch breekpunt voor gevoelig, wil zeggen dat ondanks de aanwezigheid van bepaalde resistentiemechanismen de kans op therapeutisch succes bij behandeling relatief hoog blijft (5,9).
Figuur 1.3: Escherichia coli klinische breekpunten en ECOFF voor ciprofloxacine (https://mic.eucast.org/Eucast2/Eucast2).
1.2.3 Standaardiseren van antimicrobiële gevoeligheidstesten
De drijfveer voor het standaardiseren van AST wordt ingegeven door de vele variabelen die deze methodes beïnvloeden (10). Vooral de disk diffusie methode wordt sterk beïnvloed door een aantal parameters zoals het type medium, de dikte van het medium, de inoculumconcentratie, de incubatiecondities… waardoor tijdens de eerste jaren na introductie (jaren 50) verschillende variaties van deze methode ontstonden die alleen maar tot
verwarring leidden (11). Dit gaf aanleiding tot het oprichten van een commissie in 1961 voor de ontwikkeling van AST gestandaardiseerde procedures door de Wereldgezondheidsorganisatie (10,12). De procedure werd in 1966 herbekeken en aangepast door W.M.M Kirby en A.W. Bauer, vandaag ook gekend als de Kirby-Bauer disk diffusie methode (13). In de Verenigde Staten is het CLSI (Clinical Laboratory Standards Institute) sinds 1975 verantwoordelijk voor AST richtlijnen en breekpunten. Eveneens ontstonden andere nationale commissies waardoor verschillende breekpunten voor éénzelfde antibioticum-kiem-combinatie zijn. Hierdoor werd in 1997 EUCAST (European Committee on Antimicrobial Susceptibility Testing) opgericht met als voornaamste doelen het opstellen van gemeenschappelijke breekpunten (EUCAST breekpunten) en standaardisatie van AST methodes (14,15). Laboratoria in België volgen grotendeels de EUCAST en CLSI richtlijnen op. In het algemeen liggen de waarden van de CLSI klinische breekpunten hoger dan die van de EUCAST. Dit betekent dat CLSI minder streng is dan de EUCAST doordat meer resistente bacteriën onopgemerkt blijven. De laatste jaren zien we echter ook binnen CLSI een evolutie naar lagere breekpunten (8,16).
1.2.4 Fenotypische gevoeligheidstesten 1.2.4.1 Broth dilutie methode
Het doel van een kwantitatieve bouillon of broth dilutietest is een MIC bepaling. De broth macro- en microdilutie methodes verschillen enkel in volume van het vloeibaar groeimedium, maar de macrodilutie is arbeidsintensief en daardoor niet geschikt voor routine gevoeligheidsbepalingen. De broth macrodilutiemethode wordt internationaal wel als de gouden standaard beschouwd. Een broth microdilutiesysteem bestaat doorgaans uit 96-well microtiterplaten waarvan de kuipjes gevuld zijn met een vloeibaar groeimedium en een tweevoudige verdunningsreeks van verschillende antibiotica. Er is ook steeds een well zonder antibioticum aanwezig voor de controle op groei. Aan elke well wordt een welbepaalde hoeveelheid van een 0,5 McFarland bacteriesuspensie toegevoegd. De meeste aerobe bacteriën worden 16 tot 20 uur geïncubeerd bij 35°C, terwijl anaerobe bacteriën en mycobacteriën een langere incubatieduur vereisen (24 uur of langer). Na incubatie kan visueel beoordeeld worden vanaf welke well geen groei meer waarneembaar is op basis van de troebelheid. De laagste concentratie van een antibioticum met geen visueel waarneembare groei/ troebelheid vertegenwoordigt de MIC. Sommige commerciële systemen bepalen de MIC colorimetrisch d.m.v. een kleurindicator die als reactie op bacteriële groei verandert van kleur. De laagste concentratiewell die geen kleurverandering vertoont op het moment dat de controle well verandert van kleur vertegenwoordigt de MIC (17,18).
1.2.4.2 Gradiënt diffusie test
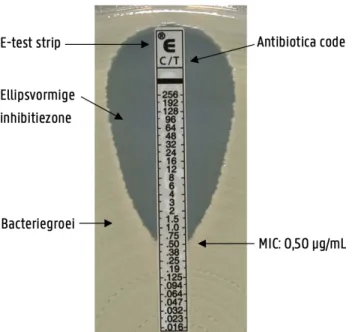
De E-test (Epsilometer test) is een andere kwantitatieve methode voor bepaling van de MIC. Een kunststofstrip, gemarkeerd met een concentratieschaal, bevat aan één kant een antibioticum
concentratiegradiënt. Wanneer de E-test op een agar, homogeen geënt met de te testen kiem, wordt gebracht, zal onmiddellijk een stabiele en continue concentratiegradiënt tot stand komen. Na overnacht incubatie ontstaat een visueel waarneembare ellipsvormige inhibitiezone en wordt de MIC afgelezen op de snijlijn van de ellips en de strip (Figuur 1.4). Deze test kan ook gebruikt worden voor trager groeiende kiemen omdat de concentratiegradiënt langdurig stabiel blijft en de E-test wordt geplaatst op specifieke groeimedia en geïncubeerd onder specifieke incubatiecondities (CO2-incubator). Het nadeel van deze methode is dat de strips duur zijn en de werkwijze arbeidsintensief is (17,18).
Figuur 1.4: E-test ceftolozane/ tazobactam met MIC 0,50 μg/mL (https://www.biomerieux-diagnostics.com). 1.2.4.3 Disk diffusie methode
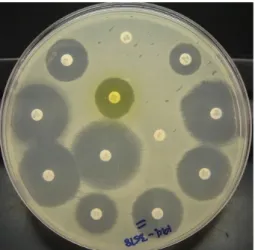
Antibioticumdisks- of schijfjes worden aangebracht op een voedingsbodem, homogeen geënt met de te testen kiem. Deze papieren schijfjes, geïmpregneerd met een concentratie antibioticum, nemen water op vanuit de agar waardoor rondom de schijfjes een concentratiegradiënt ontstaat. De methode kan enkel gebruikt worden voor snel groeiende kiemen aangezien de concentratiegradiënt slechts kortstondig stabiel is. Na ongeveer 18 tot 24 uur incubatie ontstaan inhibitiezones (Figuur 1.5). Dit zijn zones waar geen bacteriegroei optreedt door remming van de bacterie door het desbetreffende antibioticum. De diameter van de inhibitiezone (of zone diameter) is een maat voor de antimicrobiële gevoeligheid: hoe kleiner de diameter, hoe minder gevoelig de kiem. Resistente kiemen worden slechts weinig tot niet geïnhibeerd door het antibioticum waardoor ze wel kunnen groeien tot dicht bij het antibioticumschijfje.De diameter van de inhibitiezone wordt vergeleken met specifieke breekpunten van diameters, opgegeven in gestandaardiseerde tabellen van AST richtlijnen die aangeven of een kiem gevoelig (S), intermediair (I) of resistent (R) is (bijvoorbeeld EUCAST zone diameter
breekpunten). De breekpunten werden bekomen door de diameters te vergelijken met MIC-waarden, vastgelegd met de broth dilutie methode op een steekproef van bacteriën (15,19).
Figuur 1.5: Disk diffusie antibiogram met inhibitiezones (17).
1.2.4.4 MALDI-TOF MS
MALDI-TOF MS of Matrix Assisted Laser Desorption Ionization - Time Of Flight Massaspectrometrie werd oorspronkelijk geïntroduceerd voor de identificatie van micro-organismen door de generatie van een specifiek eiwitprofiel (Paragraaf 1.3.2.2). Deze toepassing kan nu ook gebruikt worden om antimicrobiële resistentie te detecteren doordat sommige vormen van resistentie, zoals bijvoorbeeld β-lactamasen, eiwitgebaseerd zijn. Een voordeel van MALDI-TOF MS is dat de identificatie en informatie over de antimicrobiële gevoeligheid op dezelfde dag beschikbaar kunnen zijn. Het gebruik ervan voor resistentiebepaling vereist wel andere voorbereidingsprotocols en een omslachtigere werkwijze dan de technieken die voor routine identificatie van micro-organismen gebruikt worden. De MALDI Biotyper® (Bruker Daltonics) en het Vitek® MS systeem (bioMérieux) zijn de twee meest gebruikte commerciële systemen (20–22). Momenteel zijn er vier verschillende methodes voor resistentiedetectie beschreven:
(i) Resistentie kan gedetecteerd worden door de analyse van verschillen tussen het spectrum van gevoelige en resistente bacteriën van dezelfde soort. Zo kunnen sommige pieken karakteristiek zijn voor resistentie. Dit is echter zeer complex en niet voor elk resistentietype mogelijk (21,22). (ii) Resistentiedetectie kan op basis van de enzymatische activiteit van bacteriën. Het Vitek® MS
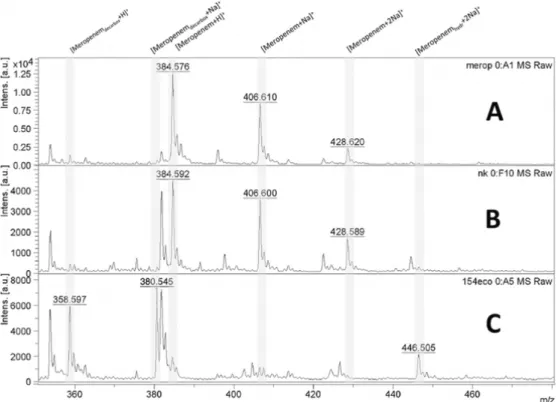
systeem beperkt zich tot nu toe tot deze benadering voor resistentiebepaling. De meest gekende en gebruikte assay is hier de MBT STAR®-BL assay (MALDI BiotyperSelective Testing Antimicrobial Resistance-Beta-Lactamase) die bacterie-geïnduceerde hydrolyse van de lactam ring van β-lactam antibiotica detecteert. De incorporatie van water zal het moleculair gewicht van het gehydrolyseerd afbraakproduct doen toenemen waardoor een waarneembare shift (+18 Da) in het
massaspectrum ontstaat. In sommige gevallen zal het product na hydrolyse worden gedecarboxyleerd (-44 Da) door de onstabiel geopende β-lactam ring. Het massaspectrum van het originele β-lactam antibioticum wordt vergeleken met het bekomen massaspectrum na 1 tot 3 uur incubatie van de bacteriesuspensie met het antibioticum. Het spectrum bestaat uit pieken representatief voor het β-lactam antibioticum, (natrium)zouten door ionisatie en/of degradatieproducten. In het geval dat hydrolyse optrad, zullen de pieken van het intacte antibioticum afnemen of zullen zelfs enkel de pieken van de degradatieproducten waarneembaar zijn (Figuur 1.6). Software berekent automatisch de intensiteitsverhouding van gehydrolyseerde en intacte antibioticumpieken, wat een maat is voor de β-lactamase activiteit (21–25).
Figuur 1.6: MALDI TOF MS spectra met pieken representatief voor meropenem, natriumzouten en degradatieproducten. (A) Meropenem oplossing (B) Negatieve controle en (C) Carbapanemase producerende
Escherichia coli (26).
(iii) De MBT-RESIST® assay (MALDI Biotyper-Resistance Test with Stable Isotopes) detecteert isotoop gelabelde aminozuren die worden opgenomen door nieuw gesynthetiseerde bacteriële eiwitten. Bacteriën worden in groeimedia met isotoop gelabelde aminozuren en in aanwezigheid van antibiotica geïncubeerd. Enkel bacteriën die kunnen groeien in de aanwezigheid van het geteste antibioticum zullen de gelabelde aminozuren incorporeren waardoor het moleculair gewicht toeneemt en dit een waarneembare piekshift in het massaspectrum veroorzaakt. Software
detecteert automatisch deze verschuivingen door het spectrum te vergelijken met het normale spectrum in afwezigheid van antibiotica (21–25).
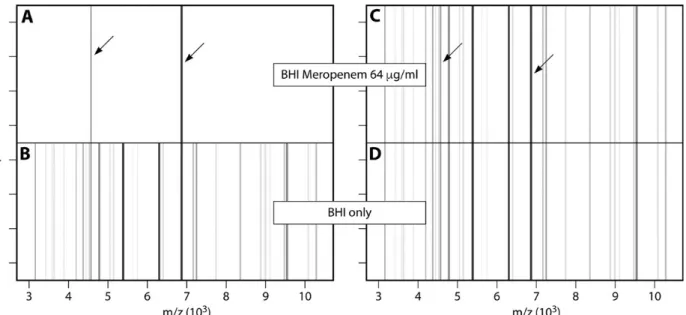
(iv) De ontwikkeling van MBT-ASTRA® (MALDI Biotyper-Antibiotic Susceptibility Test Rapid Assay) vergemakkelijkt het detecteren van kleinere eiwitten en de hoeveelheid peptiden. Net zoals bij MBT-RESIST® worden bacteriën in aan- of afwezigheid van antibiotica gekweekt, maar hier worden conventionele groeimedia gebruikt en een interne standaard toegevoegd. De piekintensiteit is gecorreleerd met de hoeveelheid bacteriële peptiden en dus ook met de bacteriegroei. Spectra van bacteriën die niet kunnen groeien in de aanwezigheid van een antibioticum en dus gevoelig zijn, zullen lage piekintensiteiten vertonen, terwijl spectra van resistente kiemen duidelijk zichtbare, intense pieken zullen hebben (Figuur 1.7). Relatieve groei wordt berekend door de verhouding van de area under curve met en zonder antibiotica. Het nadeel van MBT-RESIST® en MBT-ASTRA® is de relatief lange incubatietijd (2 tot 4 uur), die niet verkort kan worden in vergelijking tot andere MALDI-TOF MS gebaseerde resistentiedetecties (21–25).
Figuur 1.7: Massaspectra van een gevoelige (rechts) en resistente (links) K. pneumoniae na groei in het groeimedium BHI (brain heart infusion) en in aan-of afwezigheid van meropenem. Spectrum A vertoont in
aanwezigheid van het antibioticum enkel pieken van de interne standaard (zwarte pijlen) (21).
Ondanks de vooruitgang om antimicrobiële resistentie te detecteren met behulp van MALDI-TOF MS zijn er toch nog enkele beperkingen (21):
• Detectie van slechts een beperkt aantal resistentiemechanismen • Slechts een beperkt aanbod aan geautomatiseerde systemen
• Een beperkt aantal studies naar de kosteneffectiviteit van resistentiedetectie door MALDI-TOF MS
• Een gebrek aan gestandaardiseerde routines voor detectie van resistentie
• De detectie naar het effect van antibiotica op de bacteriegroei duurt langer dan andere MALDI-TOF MS gebaseerde benaderingen
1.2.5 Gentoypische gevoeligheidsbepalingen 1.2.5.1 PCR en real-time PCR
Genotypische gevoeligheidsbepalingen zijn gebaseerd op de detectie van genen die coderen voor resistentie of de aanwezigheid van mutaties die geassocieerd zijn met een verworven resistentie (5). De toenemende kennis over resistentiemechanismen en de betrokken genen maken deze benaderingen mogelijk (10). De meest gebruikte nucleïnezuur gebaseerde detectiesystemen zijn hybridisatie- en amplificatiesystemen. Beiden gebruiken de beschikbare genetische informatie in databanken voor de aanmaak van probes en primers (27).
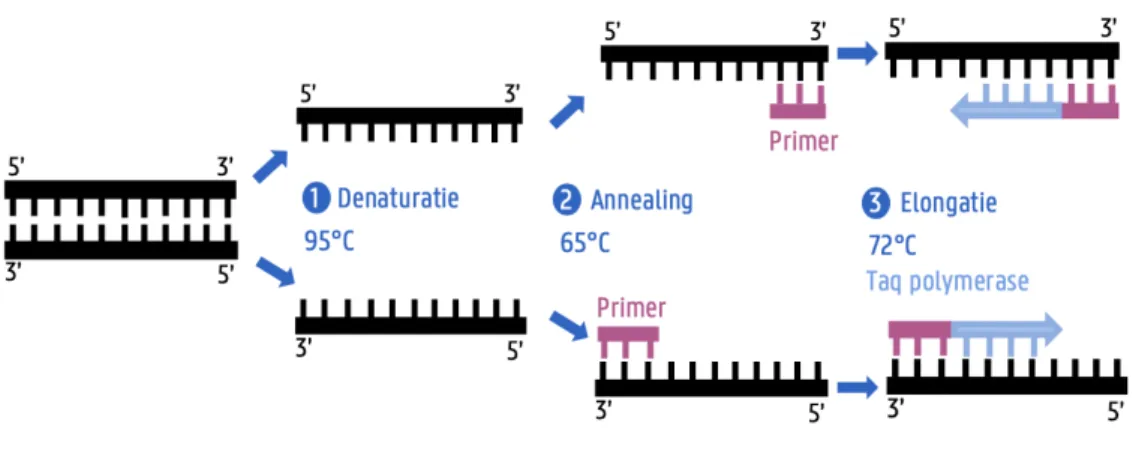
Een Polymerase Chain Reaction of PCR vermenigvuldigt exponentieel een stukje DNA zodat voldoende grote hoeveelheden worden bekomen voor een latere detectie. Een PCR cyclus bestaat uit drie stappen: denaturatie, annealing en elongatie door het DNA-polymerase (Figuur 1.8). Het DNA fragment wordt gedurende een aantal cycli geamplificeerd tot het gewenste aantal wordt bekomen. Het DNA-polymerase heeft enkelstrengig DNA nodig als template voor de synthese van een complementaire nieuwe streng. Hiervoor wordt eerst opgewarmd tot 95°C zodat dubbelstrengig DNA denatureert. Na daling van de temperatuur kunnen twee oligonucleotide primers, kleine stukjes dubbelstrengig DNA, aanhechten op een specifieke plaats van de template. De primers zijn zo gekozen dat het te amplificeren gebied omgeven wordt door een forward en backward primer. Uiteindelijk zal het DNA-polymerase een kopij van de template streng maken door complementair nucleotiden in te voegen. Het Taq polymerase, een thermostabiel DNA-polymerase, wordt gebruikt omdat het reactiemengsel telkens tot 95°C wordt opgewarmd. Bij elke PCR reactie wordt een positieve en negatieve controle meegenomen om de sensitiviteit en specificiteit te behouden. De detectie van de geamplificeerde fragmenten kan met behulp van een agarose gelelektroforese of een capillaire elektroforese gebeuren (20,28–31).
Figuur 1.8: Één PCR cyclus (eigen illustratie).
Initieel was PCR enkel een snelle identificatietool. De toenemende kennis over de genetische achtergrond van antibioticaresistentie zorgt ervoor dat PCR nu ook routinematig kan uitgevoerd worden voor de detectie van resistentiegenen, op voorwaarde dat de DNA-sequentie van het gen beschikbaar is voor aanmaak van de primers (20). Methicilline-Resistente Staphylococcus aureus (MRSA) heeft het mecA gen of een variant ervan, het mecC gen, die coderen voor een gewijzigd penicilline bindend proteïne (PBP2a) met een verminderde affiniteit voor β-lactam antibiotica. Een PCR test voor de identificatie van MRSA is gebaseerd op de detectie van mecA en mecC genen (24). Meerdere resistentiegenen detecteren is eenvoudiger geworden door een multiplex PCR reactie die het mogelijk maakt om simultaan verschillende DNA-sequenties in één reactie te amplificeren, mits gebruik van verschillende fluoroforen. De verschillende primers worden dan anders gelabeld (20,31).
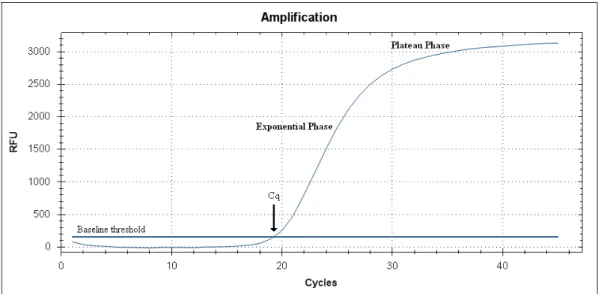
Een real-time PCR monitort de geamplificeerde producten na elke amplificatiecyclus en is daarom ook gekend als een kwantitatieve PCR. Hierdoor is detectie van de geamplificeerde producten met bijvoorbeeld gelektroforese niet nodig. Tijdsbesparing in combinatie met een kwantitatief resultaat bieden een groot voordeel (20). De productconcentratie wordt opgevolgd door middel van fluorescent gelabelde probes die hybridiseren aan hun complementaire sequentie (TaqMan probes) of intercalerende kleurstoffen die een fluorescent signaal uitzenden (SYBR Green). De fluorescentie (in Relative Fluorescene Units) wordt in functie van het aantal PCR cycli in een grafiek uitgezet, genaamd een amplificatieplot (Figuur 1.9). Pas na een aantal cycli kan het fluorescent signaal van het achtergrondsignaal of ruis onderscheiden worden. De cyclus waarbij het signaal deze drempelwaarde overschrijdt, is de Cq of kwantificatiecyclus. Vanaf deze cyclus zal het signaal exponentieel toenemen en uiteindelijk overgaan in een plateau fase. Een lage Cq is representatief voor een hoge concentratie DNA in het geamplificeerd staal (30,31). In het lab van het UZ Gent voert het GeneXpert systeem (Cepheid) een real-time PCR uit dat gebaseerd is op de detectie van resistentiegenen.
Naast het detecteren van resistentiegenen met (real-time) PCR, maakt een real-time PCR door monitoring van de geamplificeerde producten het mogelijk om de groei van bacteriën in de aanwezigheid van antibiotica op te volgen (24). Bacteriegroei in bijzijn van antibiotica wordt vergeleken met een groei controle zonder antibiotica of door het aantal veranderingen in kopijen te vergeleken met moment 0. De groei van een resistente kiem in aanwezigheid van een antibioticum wordt gedetecteerd door de toename van het aantal geamplificeerde producten gedurende een relatief korte incubatietijd van acht uur. Indirect wordt fenotypisch resistentie gemeten door groei te detecteren in de aanwezigheid van antibiotica (24,30,32).
Figuur 1.9: Een real-time PCR amplificatieplot met op de x-as het aantal PCR cycli en de y-as Relative Fluorescence Units (RFU) (30).
1.2.5.2 DNA-hybridisatie en DNA-microarray
DNA-hybridisatie is gebaseerd op de basenpaarvorming tussen purines (adenine en guanine) en pyrimidines (cytosine en thymine), namelijk adenine met thymine en guanine met cytosine. Gedenatureerd DNA wordt geïmmobiliseerd op een vaste drager, zoals een nitrocellulosemembraam. Specifieke DNA sequenties, genaamd probes, zullen hybridiseren aan het DNA zolang basencomplementariteit optreedt. Deze probes zijn gelabeld met radio-actieve isotopen, antigenen, enzymen… Na incubatie worden de niet gebonden probes weggewassen en de hybridisatiesignalen via een epifluorescentiemicroscopie gedetecteerd. Indien het gewenste gen niet aanwezig is, zullen de probes niet kunnen aanhechten en kunnen bijgevolg geen signalen worden gedetecteerd (28).
Een DNA-microarray, ook bekend als DNA-chip, is gebaseerd op een DNA-hybridisatie en kan, in tegenstelling tot een PCR reactie, tot duizenden verschillende resistentiegenen in één assay detecteren. Eerst worden probes, specifiek voor resistentiegenen, aangemaakt. Voor aanmaak van een DNA-microarray worden
deze probes gespot op een glasplaat, plastic drager of siliconen chip. Vroeger werden PCR probes gebruikt, maar tegenwoordig wordt voornamelijk gewerkt met oligonucleotide probes. In een volgende fase wordt eerst RNA geëxtraheerd, vervolgens door het reverse transcriptase omgezet naar complementair DNA (cDNA) en tenslotte fluorescent gelabeld. Één chip kan tegelijk twee monsters analyseren door aan elk een ander fluorescent label toe te kennen zodat het test- en referentie-DNA vergeleken kan worden. Vervolgens worden de gelabelde cDNA’s samen met een hybridisatie-oplossing aangebracht op de microarray zodat deze via baseparing kunnen hybridiseren aan de DNA probes. Na 14 tot 24 uur onderdompeling in een warmwaterbad van 42°C worden ongebonden cDNA’s weggewassen. De aan- of afwezigheid van resistentiegenen van de geteste kiem worden gedetecteerd door de hybridisatie resultaten te analyseren en te vergelijken met de referentie. Hiervoor scant een laserscanner de fluorescentie signalen van alle spots en wordt het gescande beeld gedigitaliseerd. Hoe intenser de fluorescentie op een bepaalde spot, hoe actiever het desbetreffende gen (20,24,28,31,33).
1.2.5.3 Whole Genome Sequencing
Voorgaande technieken, behalve DNA-microarrays, kunnen slechts een beperkt aantal resistentiegenen screenen. Multiresistente bacteriën hebben nood aan moleculaire technieken die het volledige genoom in één test kunnen onderzoeken. Next-Generation Sequencing (NGS), ter vervanging van de Sanger sequentiebepaling, biedt hiervoor een oplossing: grotere stukken DNA worden op korte tijd simultaan gesequeneerd (34). Het voordeel is dat NGS alle informatie omvat in één sequencing run: identificatie van bacteriën, gevoeligheid voor antibiotica, virulentiekenmerken en de hoeveelheid aanwezige species. Databanken bevatten informatie over de genetische factoren die resistentie bepalen. Een toepassing van NGS is Whole Genome Sequencing (WGS) dewelke een snelle én complete genoomsequenering van een bacteriestam oplevert. Louter op basis van de aanwezigheid van genen of bepaalde mutaties kan een resistentieprofiel, zonder voorkennis van het isolaats resistentie fenotype, worden opgesteld. Tevens is het ook van belang om te weten of er resistentiegenen op mobiele elementen voorkomen voor verspreidingsgevaar. In 2016 bracht de EUCAST een rapport uit met als besluit dat er enerzijds onvoldoende bewijs is dat WGS nauwkeurig antimicrobiële gevoeligheid kan bepalen en dat het anderzijds momenteel niet aan de orde is om de fenotypische AST methodes te vervangen door WGS (35). Deze conclusie volgt uit diverse redenen. WGS gebaseerde benaderingen voor AST kunnen geen MIC of zone diameter opleveren, wat meer weerspiegelt dan de aan- of afwezigheid van genen. Andere beperkingen zijn de hoge kostprijs, het gegeven dat enkel gekende resistentiegenen opgezocht worden, de moeilijkheid om databanken op te stellen die alle mogelijke genetische variaties voor resistentie omvatten en de niet altijd sterke correlatie tussen de aanwezigheid van resistentiegenen en de fenotypische resistentie. Tevens zou er op internationaal niveau een overeenstemming moeten zijn voor kwaliteitstesten en harmonisatie van benaderingen en interpretatie van de WGS voorspelde gevoeligheid. EUCAST raadt aan om deECOFF te gebruiken
als primaire vergelijking tussen de fenotypische en WGS voorspelde antimicrobiële gevoeligheid. Om een klinisch besluit te kunnen vormen over de WGS resultaten moeten de gegevens kunnen vergeleken worden met klinische breekpunten (secundaire vergelijking) (35,36). De laatste jaren zijn de kosten wel afgenomen en is de kwaliteit van WGS verbeterd. De meest gebruikte high-troughput sequencing apparaten zijn de Ion Torrent Personal Genome Machine® en Illumina® (20,36,37).
1.2.6 Fenotypische versus genotypische gevoeligheidstesten
Wanneer vroeger reeds binnen de 48 uur na ontvangst van het staal in het lab een resultaat over de antimicrobiële gevoeligheid van een kiem werd gerapporteerd, werd dit als een snel resultaat beschouwd. Nieuwe identificatietechnologieën, ter vervanging van de traditionele biochemische methodes, zoals MALDI-TOF MS en PCR maken het mogelijk om sneller resultaten te rapporteren. Een snelle identificatie is belangrijk, maar voor een optimale behandeling van infecties zijn de resultaten van gevoeligheidstesten essentieel. Ondanks dat de identificatietechnologieën steeds sneller en accurater geworden zijn, stagneerde de ontwikkeling van snelle antimicrobiële gevoeligheidstesten waardoor de impact van een vroege identificatie voor een deel teniet wordt gedaan. De laatste jaren neemt de aandacht voor snelle antimicrobiële gevoeligheidstesten daarom toe (3). Doordat fenotypische testen gebaseerd zijn op de groei van bacteriën in de aanwezigheid van antibiotica zijn resultaten pas beschikbaar na 18 tot 24 uur incubatie. In afwachting moeten patiënten een empirische antibiotische behandeling krijgen en wordt vaak gekozen voor breedspectrum antibiotica. Automatisatie kan de tijd tot 12 uur of zelfs minder reduceren, maar het zijn toch vooral de genotypische gevoeligheidstesten die het meest belovend zijn voor een snelle en nauwkeurige detectie of bevestiging van antimicrobiële resistentie. Mits continu testen inzetten, kunnen binnen de 1 tot 3 uur resultaten bekomen worden die een positief effect hebben op de klinische evolutie van de patiënt (3,10). Een bijkomend voordeel is dat sommige genotypische benaderingen de antimicrobiële gevoeligheid direct op stalen kunnen testen, bij voorkeur in combinatie met identificatie, zodat geen voorafgaande kweek nodig is (24).
Ondanks dat sommige genotypische gevoeligheidstesten veelbelovend zijn voor een snelle detectie naar de aanwezigheid van resistentiegenen blijven de fenotypische gevoeligheidstesten, en dat zal vermoedelijk ook niet snel veranderen, toch de eerste keuze bij de beslissing over de antimicrobiële behandeling bij infectie. De voornaamste redenen hiervoor zijn de flexibiliteit van fenotypische testen in combinatie met de jarenlange ervaring over de interpretatie van resultaten, de relatief lage kosten en het gemak. Deze testen laten bovendien toe om de interpretatie van de genotypische resultaten te nuanceren (3). Genotypische testen op zich hebben ook enkele beperkingen. Enerzijds betekent de afwezigheid van een resistentiegen niet dat de bacterie gevoelig is aan een welbepaald antibioticum. Anderzijds is de aanwezigheid van een resistentiegen niet noodzakelijk
gelijk aan resistentie in vitro of in vivo. De mate waarin het gedetecteerde resistentiegen tot expressie wordt gebracht moet daarom met fenotypische testen gecontroleerd worden. In sommige specifieke gevallen is er wel een sterke associatie tussen de aanwezigheid van resistentiegenen en fenotypische resistentie. Dit is het geval bij het mecA gen bij MRSA en bij de vanA en vanB genen bij Enterococcus faecium en E. faecalis (3,24,27,38). Wanneer dergelijke duidelijke correlaties bestaan, kunnen genotypische opsporingen wel de fenotypische deels vervangen. Een tweede beperking is dat een genotypische benadering enkel kan screenen naar gekende resistentiemechanismen, waardoor het continu nodig blijft om fenotypische testen uit te voeren zodat nieuw ontwikkelde resistentiemechanismen, die zich kunnen verspreiden, worden opgespoord. Verder zijn veel genotypische testen duur en is er kans op vals negatieve en positieve resultaten (24,27).
In sommige gevallen kan een moleculaire detectie superieur zijn aan fenotypische methodes zoals bij een lage in vitro expressie, heteroresistentie of een slechte groei die hogere MIC’s maskeert (27). MRSA bijvoorbeeld kan een heterogene expressie op laag niveau vertonen waardoor de fenotypische detectie moeilijker is. Daarnaast is het van belang MRSA vroegtijdig te ontdekken zodat de patiënt zo snel mogelijk geïsoleerd kan worden om de kans op transmissie naar andere gehospitaliseerde patiënten te vermijden. Hierdoor is een genotypische test, een snelle test gebaseerd op de detectie van mecA en mecC genen, de gouden standaard voor de identificatie van MRSA (27,39).
Momenteel is het niet de bedoeling om fenotypische gevoeligheidstesten te vervangen, maar om deze eerder aan te vullen met genotypische testen (3). Dit kan nuttig zijn om diverse redenen: resistentiebepaling bij traaggroeiende bacteriën, detectie naar mobiele genetische elementen verantwoordelijk voor de verspreiding van resistentiegenen, snel de aan- of afwezigheid van resistentiegenen aantonen voor een vroege interventie infectiebeheersing en ter bevestiging van specifieke resistentiemechanismen (27). MALDI-TOF MS voor resistentiebepaling combineert de voordelen van fenotypische testen met de snelheid en nauwkeurigheid van moleculaire technieken, maar vereist verder onderzoek en validatie van gegevens voor gebruik in de routine (21).
1.3 AUTOMATISATIE IN HET LABO MICROBIOLOGIE 1.3.1 Nood aan automatisatie
De hedendaags toegepaste conventionele methodes in het lab vergen vaak veel tijd en vereisen van de MLT’s veel manuele handelingen (stalen enten, incuberen, identificeren, antibiogrammen inzetten…) waardoor resultaten niet onmiddellijk beschikbaar zijn (40). Zowel de sterke toename aan te onderzoeken stalen als de afname van het aantal beschikbare MLT’s leiden tot de noodzaak aan automatisatie in het lab microbiologie. De toename van het aantal stalen is te wijten aan de toenemende levensverwachting en toenemende aandacht
voor preventie en het onder controle houden van infecties. Er zijn steeds meer immuungecompromitteerde patiënten die vatbaarder zijn voor infecties. Een bijkomende oorzaak is dat de ziekenhuizen en overheid streven naar kortere ziekenhuisverblijven waardoor snellere laboratoriumresultaten voor een patiënt worden verwacht (40,41). Daarom wordt gepoogd om met behulp van automatisatie microbiologie sneller en efficiënter te maken. De resultaten van dit proces leiden tot een aanzienlijke tijdswinst, dat mede zorgt voor een potentieel meer kosteneffectief en optimaal patiëntenbeheer (42). Door de automatisatie van eenvoudige repetitieve taken kunnen MLT’s zich focussen op andere taken waarvoor hun kennis en ervaring meer nodig is. Verder kan hierdoor het tekort aan MLT’s voor een deel worden opgevangen, anderszijds neemt het aantal gespecialiseerde taken toe waardoor ze steeds meer en diversere taken hebben. Daarnaast is een groot voordeel van automatisatie het uitsluiten van menselijke fouten zoals mislabeling, foute plaat enten, kruiscontaminatie… (43). Een belangrijk aspect van kwaliteit is de traceerbaarheid: voor elke stap die een staal ondergaat, moeten de gegevens op elk moment kunnen worden gereconstrueerd (44). De traceerbaarheid van een geautomatiseerd systeem is veel groter dan wanneer dezelfde test manueel wordt uitgevoerd (43,45).
1.3.2 Huidige automatisatie
Automatisatie in het Laboratorium voor Medische Microbiologie is globaal moeilijker tot stand gekomen dan in andere labs, zoals bijvoorbeeld in de Hematologie en in de Klinische Chemie. De verschillende staalsoorten dienen immers verzameld en getransporteerd te worden met behulp van een breed scala aan containers (steriele potjes, wissers...) en vereisen specifieke werkwijzen en incubatietechnieken waardoor automatisatie allesbehalve evident is (40,41). Bovendien zijn automaten duur en vraagt dit, net zoals in de andere labs, een reorganisatie (1,45).
In de afgelopen 10 jaar is reeds een groot deel automatisatie gebeurd in de microbiologie labs dankzij de introductie van liquid based microbiology en MALDI-TOF MS (43,45). Voorlopig is er een zekere automatisatie in het lab van het UZ Gent zoals enting door de WASP® (Copan), BactAlert (bioMérieux), PCR gebaseerde testen met GenExpert (Cepheid), identificatie met behulp van de MALDI-TOF MS (Bruker Daltonics) en AdagioTM (Biorad) voor het aflezen van disk diffusie antibiogrammen en E-testen. Voornamelijk met oog op het verkorten van de time-to-result is het tijd voor een tweede golf van automatisatie. Een laattijdige adequate antibiotische behandeling verhoogt immers de mortaliteit bij ernstige infectieziekten (38).
1.3.2.1 Liquid based microbiology
Een adequate monsterafname en een veilig transport ervan is van cruciaal belang om een kwalitatief resultaat te garanderen (1). In 2006 introduceerde het bedrijf COPAN Diagnostics een vloeistofgebaseerd
verzamel- en transportsysteem, namelijk de eSwab®. Ondertussen hebben ook andere bedrijven gelijkaardige systemen geïntroduceerd (45). De eSwab® is een combinatie van een flocked swab en een vloeibaar Amies transportmedium. De flocked swab wordt aangemaakt door duizenden korte nylonvezels met behulp van een capillaire aanzuigkracht aan het oppervlak van een staafje te brengen waardoor deze meer staal kan opnemen en afgeven dan een klassieke wisser. Dankzij het vloeibaar transportmedium is het staal beschikbaar voor enting door een automaat en kunnen er meerdere testen op één staalname worden uitgevoerd. Het medium is tevens ideaal voor aerobe, anaerobe en moeilijk groeiende bacteriën (1,44–46).
Vroeger was het noodzakelijke stalen voornamelijk manueel te enten op media, omdat stalen met een hogere viscositeit (vaste en halfvaste stalen) automatisatie bemoeilijkten. Sinds de introductie van een liquid
based microbiology kan deze taak in het lab voornamelijk overgenomen worden door entautomaten. De meest
gekende entautomaten zijn: de Autoplak® (Beckman Coulter), de WASP® (Copan Diagnostics), de PreLUD® (I2A Diagnostics), de Inoqula® (Becton Dickinson Kiestra) en de Previ-Isola® (bioMérieux). Het belangrijkste verschil tussen deze automaten is de manier waarop platen worden geënt: Autoplak®, WASP® en PreLUD® enten met een hitte gesterilliseerd entoog, Inoqula® met magnetische kogeltjes en Previ-Isola® met een kammetje (1,45,47,48). Geautomatiseerd enten heeft tal van voordelen t.o.v. manueel enten: een betere traceerbaarheid, geen kruiscontaminatie, minder menselijke fouten... Het lab van het UZ Gent gebruikt de WASP® voor automatische enting van stalen. De operator moet enkel de stalen en de media plaatsen en kan daarna letterlijk –gedurende een bepaalde tijd- weggaan (‘Walk Away!’) om andere taken uit te voeren. Dankzij de koppeling met het LIS (Laboratorium Informatica Systeem) worden met behulp van gegevens uit het LIS onmiddellijk de juiste platen geënt. Wanneer er te weinig staal voorhanden is of in geval van specifieke stalen, zoals kathetertips die niet vloeibaar te maken zijn en dus niet door de WASP® kunnen worden geënt, gebeurt de enting wel manueel (47).
1.3.2.2 Identificatie met MALDI-TOF MS
De identificatie van micro-organismen gebeurt meestal met behulp van MALDI-TOF MS. Een laserpuls doet ionen ontstaan die versneld worden in een elektrisch veld en daarna worden gescheiden op basis van hun massa/ladingsverhouding (m/z). Het resultaat is een uniek massaspectrum waarbij de pieken de verschillende m/z waarden van de gevormde ionen en de hoogte de intensiteit weergeven (49). Het bekomen massaspectrum wordt automatisch vergeleken met een referentiespectrum en afhankelijk van het aantal overeenstemmende pieken wordt een identificatiescore toegekend (22,50).
1.3.3 Total Laboratory Automation
De implementatie van een Total Laboratory Automation (TLA) waarbij automaten voor inoculatie, incubatie, beeldvorming en aflezing d.m.v. transportbanden (track) zijn gekoppeld, biedt veel voordelen voor het lab. Eerst worden stalen automatisch geënt op media om deze vervolgens via de track te verplaatsen naar de incubator. Na een gedefinieerde incubatieduur en verplaatsing naar het image acquisition station wordt een digitaal beeld met een hoge resolutie vastgelegd voor de automatische detectie van groei. Dit wordt vergeleken met een digitaal beeld, genomen op tijdstip 0 (bij start van de incubatie). Groei kan hierdoor vroegtijdig worden opgespoord en de incubatieduur verkorten. MLT’s bekijken de digitale beelden en beslissen over het verdere verloop (verder incuberen, weggooien, verdere manuele verwerking…). Dit biedt tal van voordelen: minder kans op menselijke fouten en contaminatie, opgeslagen beeldanalyses, een optimale traceerbaarheid, arbeidsbesparing en door gedigitaliseerde beelden kan de aflezing ook buiten het lab gebeuren. De twee beschikbare systemen van totale labautomatisatie zijn Becton Dickinson’s Kiestra TLA® en Copan’s WASPLab®, respectievelijkgekoppeld aan Inoqula® en WASP® voor enting. Recent ontwikkelden deze bedrijven automaten die kolonies oppikken, enerzijds voor de identificatie met MALDI-TOF MS en anderzijds voor de aanmaak van bacteriesupensies voor de disk diffusie methode (40,41,45,51).
1.4 ENTEN MET DE WASP® in UZ Gent
Figuur 1.10: Vooraanzicht van de WASP® (https://www.copanusa.com).
De WASP®, ontwikkeld door het bedrijf COPAN Diagnostics, is een entautomaat die sinds 2013 in het UZ Gent wordt gebruikt (Figuur 1.10). Enkel vloeibare stalen kunnen worden geënt (of geïnoculeerd) met de WASP® waardoor staalvoorbereiding van cruciaal belang is. Aan faecesstalen, die niet vloeibaar genoeg zijn, wordt 1 mL steriel, fysiologisch water toegevoegd vooraleer ze door de WASP® geënt kunnen worden. Verder beschikt de WASP® over een GramSlidePrep module die uitstrijkjes aanmaakt en labelt voor de Gramkleuring (44). Daarnaast
is er ook plaats voor een disk- en brothcarrousel die automatisch met een dispenser antibioticumschijfjes voor de disk diffusie op platen aanbrengt (Figuur 1.11). De dispensing disk heeft tevens de mogelijkheid om een optochine schijfje aan te brengen zodat dit ook niet meer manueel hoeft te gebeuren. De broth inoculatie module inoculeert automatisch een aanrijkingsmedium voor stalen. De aanrijkingsmedia worden op de halfronde pallet van de carrousel geplaatst (Figuur 1.12). Bijvoorbeeld een thioglycolaat bouillon is een specifiek medium voor moeilijk en traaggroeiende anaerobe bacteriën (1). De disk- en brothcarrousel zijn niet geïnstalleerd op de WASP® van het UZ Gent.
Figuur 1.11: Disk carrousel van de WASP® (1).
De WASP® bestaat uit twee robot-armen, namelijk Tarzan voor de verplaatsing van stalen en Jane voor enting (Figuur 1.12). Verschillende stalen, elk met een specifieke barcode, worden geladen op een laadplatform. De WASP® werkt LIS-gekoppeld waardoor staal- en patiëntengegevens uitgewisseld kunnen worden. Na scanning van de barcode zal Tarzan het staal overbrengen naar de vortex. Afhankelijk van het protocol selecteert de WASP één van de drie soorten entogen: 1 µL, 10 µL of 30 µL. Entoog 1 µL wordt gebruikt voor urinestalen, entoog 10 µL voor respiratoire stalen en entoog 30 µL voor de varia stalen zoals wondvochten, uitstrijkjes, brandwonden, etters... Vanuit de carrousel worden het juiste medium aangevoerd en geïnoculeerd met het juiste entoog. In de carrousel is er plaats voor negen verschillende types media met telkens veertig platen. Ondertussen zal Tarzan het volgende staal naar de vortex verplaatsen. Na elke enting wordt het entoog gesteriliseerd in de heating block. Een camera verifieert steeds of het entoog voldoende monster bevat. Wanneer dit niet het geval is, zal het entoog terug in het staal gaan. Indien na drie pogingen dit niet lukt, wordt het staal geweigerd en zal Tarzan deze verplaatsen naar het uitwerpbakje. Na enting wordt een etiket aangebracht en zal Tarzan de plaat verplaatsen naar de ‘sort out carrousel’ (een pre-incubator) (1,44).
Er zijn vier opties: • CO2: • Other • 35°C • To do
Soms is wel nog een manuele handeling nodig. Voor de identificatie van Streptococcus pneumoniae bijvoorbeeld wordt een schijfje optochine manueel aangebracht op de platen.
Figuur 1.12: Bovenaanzicht van de WASP® met (1) robotarm Jane (2) robotarm Tarzan en (3) de disk- en brothcarrousel (https://www.copanusa.com).
1.5 AUTOMATISATIE ANTIMICROBIËLE GEVOELIGHEIDSTESTEN
Zoals eerder vermeld (Paragraaf 1.2.6) zijn fenotypische gevoeligheidstesten gebaseerd op de detectie van bacteriegroei in de aanwezigheid van antibiotica waarvoor lange incubatietijden (18 tot 24 uur) worden vereist vooraleer interpretatie mogelijk is. Automatisatie reduceert de benodigde tijd voor uitvoering van deze testen. Sommige geautomatiseerde systemen kunnen zelfs de incubatieduur verkorten door vroegtijdig kleine veranderingen in bacteriegroei te detecteren, wat meer impact heeft op de time-to-result. Een voorafgaande kweek blijft wel nog steeds nodig en is een tijdrovende factor (10,52).
1.5.1 Systemen voor de disk diffusie methode
Verschillende parameters van de disk diffusie methode (type medium, inoculum densiteit…) werden gestandaardiseerd. Het meten van de zone diameters door MLT’s is een subjectieve waarneming dat zorgt voor een operator afhankelijke variabiliteit. Automaten voor aflezing van de zone diameters, zoals AdagioTM (Biorad) in het lab van het UZ Gent, zijn dan ook een nuttig hulpmiddel voor de standaardisatie van deze variabele. Een geautomatiseerde zone diameter lezer neemt een foto van antibiogrammen, meet automatisch de diameters van de inhibitiezones en interpreteert deze met behulp van de gekoppelde software naar S, I of R. Ondanks het feit dat manueel nazicht en kleine correcties noodzakelijk blijven, neemt het aflezen met behulp van AdagioTM in
ieder geval minder tijd in beslag dan het manueel meten en interpreteren van elke diameter en is er tevens een veel kleiner risico op fouten. Andere hulpsystemen voor de disk diffusie methode zijn een disk dispenser om antibioticumschijfjes aan te brengen en turbidimetrische apparaten om bacteriesuspensies met een 0,5 McFarland densiteit te bekomen (11,52,53). Een volledige automatisatie van de disk diffusie methode kan door koppeling van de WASP® aan het WASP®Lab, zoals aangehaald in Paragraaf 1.3.3. De WASP® brengt na enting van een bacteriesuspensie antibioticumschijfjes aan zodat daarna het antibiogram via de transportband verplaatst wordt naar het WASPLab® voor een verdere verwerking (41).
1.5.2 Systemen voor de broth microdilutie
Verschillende geautomatiseerde hulpsystemen voor de broth microdilutie zijn beschikbaar. De Sensititre AutoInoculator® (Trek Diagnostics) bijvoorbeeld inoculeert automatisch microtiterplaten met bacteriesuspensies. Afleessystemen, zoals Sensititre Autoreader® en AutoSceptor® (Becton Dickinson Diagnostics), evalueren automatisch bacteriegroei in microtiterplaten. Afhankelijk van het systeem is de detectie gebaseerd op fotometrie, turbidimetrie of fluorimetrie (52).
1.5.3 Geautomatiseerde broth-gebaseerde gevoeligheidstesten 1.5.3.1 Historiek
Het Autobac® disk elutie systeem (Pfizer Diagnostics) was één van de eerste automaten die het effect van een antibioticum op de ontwikkeling van bacteriën in een bouillon waarneemt op basis van lichtverstrooiing (54). Een test cuvette bestaat uit dertien kamers waarvan twaalf kamers voor het testen van antibiotica en één controlekamer zonder antibioticum. Elke kamer wordt gevuld met een bepaalde hoeveelheid bacteriesupensie. Antibioticumschijfjes worden boven op de diverse kamers geplaatst zodat deze contact maken met de bouillon, maar zonder binnen te dringen in de kamer en hierdoor niet het licht verstoren. De kamers worden tijdens incubatie bij 35°C geschud zodat het antibioticum uit de schijfjes elueert in de kamers. Een lichtverstrooiende fotometer evalueert de hoeveelheid groei in elke kamer en vergelijkt die met de groei in de controlekamer. Een groei index tussen 0 en 1 wordt bekomen waardoor de gevoeligheid voorspeld kan worden (52,54).
Het Abbott MS-2® systeem, ontwikkeld in 1977, is vergelijkbaar met de Autobac®. Een fotometer neemt turbidimetrische en colorimetrische veranderingen waar in de bouillon waarin het micro-organisme groeit (54). Datzelfde jaar introduceerde McDonnell Douglas Corporation het Automicrobic® systeem, de voorloper van Vitek®, dat gebruik maakt van gedehydrateerde reagentia in plastieken testkaarten, dewelke beschikbaar zijn voor identificatie en gevoeligheidstesten van verschillende micro-organismen. De implementatie van gestandaardiseerde microtiterplaten met antibiotica, ook ontwikkeld in 1977, speelde een belangrijke factor bij
de ontwikkeling van geautomatiseerde gevoeligheidstesten (10). Dit leidde tot het ontstaan van SensititreTM, BBL Sceptor®, een reeks systemen van Microscan en evolueerde verder naar meer complexere systemen zoals AutoScan® en het MicroScan Walkaway® systeem. Van de verschillende systemen zijn er slechts vier die het meest worden gebruikt: Vitek®, SensititreTM, MicroScan WalkAway® systeem en BD PhoenixTM (vergelijking in Tabel 1.1). Het voordeel is dat ze de benodigde tijd vooraleer resultaat kunnen reduceren, arbeidsbesparend en eenvoudig zijn. Nadelen zijn de kostprijs, het optreden van onzuiverheden, de beperkte flexibiliteit met voorgemaakte microtiterplaten en moeilijkheden om heteroresistentie en gemengde culturen te detecteren (10,52).
1.5.3.2 Vitek® en Vitek® 2 systeem
Het Vitek® systeem (bioMérieux) is een geautomatiseerd systeem dat naast het uitvoeren van een antimicrobiële gevoeligheidstest ook snel micro-organismen kan identificeren. Een identificatiekaart bestaat uit 30 wells gevuld met groeimedia en identificeert bacteriën d.m.v. enzymatische reacties. Een gevoeligheidskaart bestaat uit 45 wells gevuld met groeimedia en verschillende concentraties antibiotica. Één well zonder antibiotica dient als positieve controle zodat groei in wells hiermee vergeleken kan worden en er rekening gehouden wordt met de groeisnelheid. Vitek® beschikt over een vulsysteem dat elke well inoculeert met een 0,5 McFarland bacteriesuspensie, een incubatie- en afleessysteem en een computersysteem. Om de vijftien minuten worden turbidimetrische metingen uitgevoerd om bacteriegroei in de wells te detecteren. De gemeten transmissie is omgekeerd evenredig met de totale biomassa van de bacteriën. De verandering van de transmissie (uitgedrukt in %) wordt ten opzichte van de tijd uitgezet in een curve (Figuur 1.13). De MIC wordt bepaald door de curve te vergelijken met databanken die standaardcurves met gekende MIC’s omvatten (geëxtrapoleerde MIC’s) (52,55).
Een meer geautomatiseerde versie is het Vitek® 2 systeem (bioMérieux), beschikbaar in drie formaten die verschillen in capaciteit en automatisatie: Vitek 2 compact, Vitek 2 en Vitek 2 XL. Kaarten worden automatisch na inoculatie naar het incubatie- en afleessysteem gebracht en op het einde weggeworpen. Een 64-well kaart die tot wel twintig antibiotica kan testen wordt, net zoals bij Vitek®, om de vijftien minuten turbidimetrisch gemonitord op groeiactiviteit (17,52).
Figuur 1.13: % Verandering transmissie in functie van de tijd met de blauwe lijn als positieve controle en de groene, gele en rode lijnen antibioticum concentratie (in μg/mL) (https://www.biomerieux-microbio.com).
1.5.3.3 SensititreTM ARIS
SensititreTM Automatic Reading and Incubation System (ARIS, Thermo Scientific) is een geautomatiseerd overnacht incubatie- en afleesssysteem, ontwikkeld in het jaar 1980. Gestandaardiseerde 96-well microtiterplaten worden na innoculatie met de Sensititre Autoinoculator® in de incubator geplaatst. Bacteriegroei wordt fluorimetrisch gemonitord door hydrolyse van fluoroforen. Fluoroforen bestaan uit substraten gelinkt aan fluorescerende componenten, zoals het 4-methylumbelliferone. Deze complexen fluoresceren normaal niet, maar door bacteriegroei ontstaan specifieke enzymen die fluoroforen hydrolyseren. Hierdoor zullen de fluorescerende componenten, nu losgekoppeld van de substraten, fluoresceren onder UV-licht. Gevoelige bacteriën kunnen weinig of niet groeien in aanwezigheid van antibiotica waardoor geen fluorescentie waarneembaar zal zijn. Computer software rapporteert MIC’s (17,52).
1.5.3.4 MicroScan WalkAway® systeem
Het MicroScan WalkAway® systeem (Beckman Coulter) maakt gebruik van gestandaardiseerde 40- of 96- well microtiterplaten die manueel worden geïnoculeerd en daarna in het systeem worden geplaatst voor incubatie en analyse. Platen worden overnacht of een verkorte tijd geïncubeerd met respectievelijk een fotometrische en fluorimetrische detectie van bacteriegroei (17).
1.5.3.5 BD PhoenixTM
Het Becton Dickinson’s PhoenixTM paneel is een zelf-inoculerend polystyreen tray met 136 wells. Het paneel bestaat uit 51 wells gevuld met gedroogde biochemische substraten voor identificatie van bacteriën en 85 wells, waarvan één controle well, voor een antimicrobiële gevoeligheidstest met telkens een tweevoudige verdunningsreeks van elk antibioticum. Het PhoenixTM systeem detecteert bacteriegroei met behulp van een redox-indicator. Bacteriën die in de aanwezigheid van antibiotica kunnen groeien, leiden tot een kleuromslag van de indicator. Om de twintig minuten gebeurt een colorimetrische meting en een turbiditeitsmeting (55).
Tabel 1.1: Vergelijking automatische systemen.
Systeem Capaciteit Principe Resultaat AST
Vitek® 2 (bioMérieux) 64-well kaart met 1 - 6 concentraties van 9 - 20 antibiotica Turbidimetrische meting om de 15 minuten 4 – 10 uur SensititreTM ARIS (Thermo Scientific)
96-well microtiterplaten Fluorimetrische meting door hydrolyse van fluoroforen
18 - 24 uur MicroScan WalkAway® (Beckman Coulter) 40- of 96-well microtiterplaten
Verkorte incubatie: detectie fluorimetrisch Overnacht incubatie: detectie
fotometrisch
3,5 - 7 uur
18 uur of langer
BD PhoenixTM Polystyreen tray met 136
wells waarvan 85 wells voor AST
Colorimetrische meting (redox-reactie) en turbiditeitsmeting om
de 20 minuten
2. OBJECTIEVEN
Zowel de steeds meer toenemende resistentieproblematiek als het aantal te testen stalen zorgen ervoor dat dagelijks talrijke antimicrobiële gevoeligheidstesten worden ingezet om voor een patiënt een zo gericht mogelijke antibiotische behandeling te verzekeren. MLT’s besteden veel tijd aan deze soms routineuze taak, terwijl er diverse andere taken, die meer expertise vereisen, zijn. Verschillende geautomatiseerde antimicrobiële gevoeligheidstesten kunnen reeds een aanzienlijke tijdswinst opleveren, maar automatisatie van de meest gebruikte fenotypische methode, de disk diffusie methode, is beperkt. Deze methode is momenteel semi-geautomatiseerd in het Laboratorium voor Medische Microbiologie van het UZ Gent: alle handelingen worden manueel uitgevoerd, behalve aflezing van de diameters van de inhibitiezones gebeurt d.m.v. het semi-geautomatiseerd systeem AdagioTM (Biorad). Automatisch diameters aflezen, ter vervanging van manuele metingen, leidt tot een betere reproduceerbaarheid, precisie en tijdswinst (56). Deze automatisatie biedt ook een zekere standaardisatie van de variabele. Andere factoren die AST variatie veroorzaken, zoals de inoculatie van bacteriesuspensies, zijn moeilijker om te standaardiseren. Het doel van dit onderzoek is om na te gaan of een automatische enting van Mueller-Hinton of Mueller-Hinton Fastidious platen voor disk diffusie antibiogrammen door de entautomaat WASP® accuraat en efficiënt is.
Concreet wordt de conventionele, manuele disk diffusie methode vergeleken met antibiogrammen geënt door de WASP®. Na enting door de WASP® worden manueel de antibioticumschijfjes aangebracht zodat daarna de antibiogrammen kunnen worden geïncubeerd. Automatisatie moet de resultaten van de manuele werkwijze minstens evenaren of zelfs verbeteren. De kans bestaat dat bij vergelijking van de twee methodes, met de manuele methode beschouwd als referentie, discrepanties optreden. Maximale percentages van discrepanties (minor, major of very major errors) mogen niet overschreden worden om te besluiten of de methodes vergelijkbare resultaten opleveren. Daarnaast is ook de tijdsinvestering van belang. Voor toepassing in de routine zou deze methode evenveel tijd als de conventionele methode moeten vergen of nog idealer zelfs een tijdwinst opleveren. Via een interpersoneelsvergelijking wordt vergeleken in welke mate MLT’s dezelfde resultaten bekomen bij aflezing van de antibiogrammen met AdagioTM.
3. MATERIALEN EN METHODEN
3.1 PATROON EN ENTOOG
3.1.1 Materialen
• Buisjes met steriel, fysiologisch water LOT 06/02/2020
• Twee stammen: Staphylococcus aureus ATCC 29213 en Streptococcus pneumoniae ATCC 49619 • Steriele swabs LOT 1819
• Plastieken buisjes (Copan, Brescia, Italië) • Entogen 10 μL en 30 μL (Copan, Brescia, Italië)
• WASP® (Copan, Brescia, Italië) en WASP®-rekjes (Copan, Brescia, Italië) • Mueller-Hinton agar LOT64310663 (Biorad, Marnes-la-Coquette, Frankrijk)
• Mueller-Hinton Fastidious agar LOT64323574 (Biorad, Marnes-la-Coquette, Frankrijk)
• Antibioticumschijfjes voor S. aureus (Biorad, Marnes-la-Coquette, Frankrijk): rifampicine 5 μg (RA), cefoxitine 30 μg (FOX), tetracycline 30 μg (TE), clindamycine 2 μg (CM), linezolid 10 μg (LZD), trimethoprim/sulfamethoxazole 25 μg (SXT)
• Antibioticumschijfjes voor S. pneumoniae (Biorad, Marnes-la-Coquette, Frankrijk): moxifloxacine 5 μg (MXF), vancomycine 5 μg (VA), ampicilline 2 μg (AM), benzylpenicilline 1 IU (International Unit) (P1), clindamycine 2 μg (CM) en oxacilline 1 μg (OX)
• Incubator op 35°C (Binder, Tuttlingen, Duitsland) • CO2-incubator op 35° (Memmert, Leuven, België) • AdagioTM (Biorad, Marnes-la-Coquette, Frankrijk)
3.1.2 Methoden
Dit experiment beschrijft welk patroon en welk entoog op de WASP® de beste resultaten geven voor de enting van disk diffusie antibiogrammen. Verschillende protocollen zijn geprogrammeerd op de WASP® voor enting van twee ATCC stammen:
Protocol 1:
- Medium: Mueller-Hinton (MH) agar - Entoog: 10 μL
- Entpatroon: x2 (Figuur 3.1): de helft van de plaat wordt geënt, daarna gaat het entoog terug in het staal en ent de andere helft van de plaat (10 μL x2)