huidmondjes bij planten
Een universeel model voor het detecteren van
Academiejaar 2019-2020
Master of Science in de industriële wetenschappen: elektronica-ICT Masterproef ingediend tot het behalen van de academische graad van
Begeleider: ir. Olivier Pieters
Promotor: prof. dr. ir. Francis wyffels
Studentennummer: 01601744
Arthur Van de Velde
huidmondjes bij planten
Een universeel model voor het detecteren van
Academiejaar 2019-2020
Master of Science in de industriële wetenschappen: elektronica-ICT Masterproef ingediend tot het behalen van de academische graad van
Begeleider: ir. Olivier Pieters
Promotor: prof. dr. ir. Francis wyffels
Studentennummer: 01601744
Arthur Van de Velde
Dankwoord
In dit dankwoord wil ik graag zeer uitdrukkelijke mijn promotor professor Francis wyffels en assistent Olivier Pieters bedanken. Dankzij hun grote hulp is het gelukt om deze masterproef binnen het eerste semester af te ronden.
Door de speciale omstandigheden gedurende het tweede semester van 2020 ben ik met een nieuwe masterproef moeten beginnen rond midden maart. De enige rede dat het toch gelukt is om in eerste zit in te dienen is dankzij mijn promotor prof. Francis wyffels en assistent Olivier Pieters. Midden maart ben ik noodgedwongen op zoek moeten gaan naar een nieuwe masterproef. Zij hebben mij zeer snel geholpen aan een interessant onderwerp. Zo was het mogelijk om direct van start te gaan. Maar ik wil hen vooral bedanken om elke week met mij te skypen. Er werd grondig geluisterd naar wat ik had bereikt en er werd direct bijgestuurd waar nodig. Deze skype gesprekken konden gemakkelijk meer dan een uur duren wat echt toont dat er oprechte interesse was en goede hulp geboden kon worden. Ik besef goed dat niet elke masterproef student het geluk heeft om zo een goede promotor en assistent te hebben. Hiervoor wil ik hun dan ook uit de grond van mijn hart bedanken.
Zonder de data was het niet mogelijk om deze masterproef tot een goed einde te brengen. Daarom wil ik graag PhD student Sofie Meeus bedanken. Zij heeft de data aangeleverd en heeft alle huidmondjes reeds gelabeld. Zelf ben ik nog eens over deze labeling gegaan en ik besef dus goed wat een titanen werk dit is.
Verder zou ik graag nog professor Verhaevert bedanken. Hij heeft mede gezorgd voor een snelle overgang naar een nieuw onderwerp voor deze masterproef. Het goede contact met prof. Verhae-vert heeft heel veel geholpen in het begin van deze masterproef. Ook de planning voor andere vakken werd vlot geregeld waardoor ik direct met volle focus kon beginnen.
Ook wil ik graag nog mijn vader bedanken om al mijn geschriften na te lezen en te verbeteren waar nodig. En verder zou ik graag nog mijn broer en zussen en mijn moeder bedanken voor de goede en vlotte thuissituatie. Het belang hiervan is in deze corona situatie niet te onderschatten. Tot slot wil ik graag de mensen van het GPU lab bedanken. Zij maakten het mogelijk om vlot te werken met de servers van de UGent. De vlotte en heldere communicatie met hen heeft vele uren frustratie kunnen uitsparen.
Bedankt allemaal! Arthur Van de Velde
Toestemming voor gebruik van inhoud
De auteur(s) geeft (geven) de toelating deze masterproef voor consultatie beschikbaar te stellen en delen van de masterproef te kopiëren voor persoonlijk gebruik. Elk ander gebruik valt onder de bepalingen van het auteursrecht, in het bijzonder met betrekking tot de verplichting de bron uitdrukkelijk te vermelden bij het aanhalen van resultaten uit deze masterproef.
COVID-19-preambule
De eerste maanden van 2020 zullen niet snel vergeten worden. Dit waren namelijk de maanden waarin de wereld werd getroffen door COVID-19. Op het moment van de uitbraak was ik op Erasmus in Grenoble (Frankrijk). Door deze speciale omstandigheden was het niet aangeraden om daar te blijven. Bijgevolg is er gekozen om terug gekomen en de studies af te werken in Gent. Dit was op het moment zelf een moeilijke beslissing maar achteraf zeker de juist beslissing. Door de terugkomst was het nodig om een nieuwe masterproef te beginnen. De overgang ging vlot en het is gelukt om begin maart te beginnen. Dit is natuurlijk later dan de geplande start datum van een masterproef.
Gelukkig was er zeer goede ondersteuning, door de wekelijkse meetings en snelle communicatie is het gelukt om goed door te werken. Er is ook gekozen voor een onderwerp dat volledig software gericht is om zo moeilijkheden met samenkomsten te vermijden. Door deze keuze kon alles van thuis uit gebeuren.
Toch waren dit nog steeds speciale omstandigheden die de hele procedure niet hebben vereenvou-digd. Zo was het moeilijker om met medestudenten vragen te bespreken. Van thuis uit werken ging redelijk vlot maar was niet altijd even eenvoudig. Al bij al is de hinder beperkt gebleven door de vlotte communicatie. Maar dit heeft het werk zeker niet vereenvoudigd.
Abstract
De toekomst zit in neurale netwerken. Door de vooruitgang in computer kracht van de laatste jaren is er ook een grote vooruitgang in de neurale netwerken. Door deze vooruitgang zijn neurale netwerken nu in staat om objecten op een afbeelding te herkennen. Dit is zeer nuttige voor allerlei toepassingen.
Huidmondjes worden door biologen onderzocht om
Biologen doen onderzoek op huidmondjes bij planten. Huidmondjes bevatten tal van informatie. Het is bijvoorbeeld mogelijk voor biologen om de CO2 concentratie in de atmosfeer op te volgen aan de hand van huidmondjes. Om deze onderzoeken te verrichten is het nodig de huidmondjes te detecteren op bladeren. Dit is momenteel een zeer duur en tijdrovend proces.
In deze paper wordt onderzocht hoe het detecteren van huidmondjes automatisch kan verlopen. Om dit te bekomen wordt er gebruik gemaakt van een Faster R-CNN netwerk. Dit is een neuraal netwerk dat werd ontwikkeld voor object detectie. Er wordt gekeken naar de nodige data en hoe het netwerk geoptimaliseerd wordt. Uiteindelijk worden de resultaten besproken.
Trefwoorden Faster R-CNN, Neurale netwerken, Deep learning, Huidmondjes, Object detectie
1
A universale model for the detection of
stomatat in plants
Van de Velde Arthur
Supervisor: prof. dr. ir. Francis wyffels Counsellor: ir. Olivier Pieters
Ugent
ABSTRACT
Abstract—The future is in neural networks. The progress in computer power of the last few years made the way for great progress in neural networks. Because of this progress, neural networks are now able to recognize objects in an image. This is very useful for all kinds of applications.
Biologists do research on stomata in plants. Stomata contain a lot of information. For example, it is possible for biologists to monitor the CO2concentration in the atmosphere using stomata.
To perform these studies it is necessary to detect stomata on leaves. This is currently a very expensive and time-consuming process.
This paper investigates how the detection of stomata can be done automatically. To obtain this, a Faster R-CNN network is used. This is a neural network that was developed for object detection. The data needed to train the network will be examined. After this the network will be optimized and the results will be investigated.
Index Terms—Faster R-CNN, Neural network, Deep learning, Stomata, Object detection
I. INTRODUCTION
Plant organisms are capable of building up their own carbon compounds. Plants will extract carbon dioxide (CO2) from the
atmosphere to build up these carbon compounds. The removal of CO2from the atmosphere is done through stomata. Stomata
are present on the leaves of a plant. The stomata are able to open and close, when they are open CO2is extracted from the
atmosphere.
The formation of stomata on the leaves of plants will depend on both short and long term conditions. For example, a leaf formed during a cold and wet winter will be large and will contain a lot of stomata. This while leaves formed in a dry and warm summer are rather small and contain little stomata [1]. The formation of stomata will also depend on long-term environmental factors. For example, plants that contain leaves with a lot of stomata will have a greater chance of survival when the CO2 concentration in the air drops. Conversely,
plants with few stomata will have a greater chance of survival when the CO2 concentration in the atmosphere rises [1].
On the basis of these long-term effects it is possible for biol-ogists to monitor the CO2concentration in the atmosphere and
monitor its change. For example, the 400 million years during the Phanerozoic era, associated with a low CO2concentration,
are characterized by leaves with a high stomata concentration. Detecting stomata on leaves is therefore not unimportant for researchers. Currently, this is done manually, which makes
it an expensive and time-consuming undertaking. However, thanks to recent advances in neural networks, it is possible to automate this process. The aim of the master thesis is to develop a model that is able to detect stomata on leaves. This model is not plant-specific but should work well on different plant species. To achieve this goal a Faster Region-based Convolutional Neural Network (Faster R-CNN) is used.
In part 2 the data obtained and how it is processed to train and test the neural network will be discussed in more detail. Part 3 explains how the Faster R-CNN network works. Part 4 examines the metrics. In part 5 the parameters are listed with their optimal value. In part 6 the results are reviewed. In part 7 the implementation is briefly mentioned and in part 8 everything is summarized.
II. DATA EXPLORATION AND PREPROCESSING
A first and important step in any neural network is the data. Without data it is not possible to train a neural network and without the correct data this training will go nowhere. In this section we will go deeper into the data used and how this data has been processed to train the neural network correctly.
A. Used data
The neural network aims to detect stomata on images of plants. To train the network for this, images of stomata on different plants are needed. An example of such an image is shown in figure 1. The complete data set consists of 113 different plant species combined with a .txt file on which the locations of all stomata are given. The .txt file contains the name of the plant species, combined with the x and y coordinates of the stomata and the size of the stomata.
The original data consists of RGB images of 1600 by 1200 pixels. These images were taken manually with a microscope. It is possible that there are differences between the images. For example, an image may be overexposed or underexposed. It is also possible that some images are less sharp or even blurred. Both fresh and dried materials are used. This can result in a difference in color. The fresh material will be greener than the dried material. Some plant species have an orange glow which differs from the green of most other plants. To deal with all these differences data preprocessing has been used.
2
Figure 1: Example image of the data.
B. Generating data
An original image is shown in figure 1. This is an RGB color image of 1600 by 1200 pixels. For the neural network, images of 800 by 600 pixels are used. When generating data, it is therefore important to crop each image to 800 by 600 pixels. This can be done by cutting the image into four pieces.
It is also important that the model receives an equal amount of data for each plant species. Therefore, 200 stomata are generated for each plant. The original data includes plants for which sufficient images are available. With these plants it is therefore not necessary to generate extra data. Other plants only have a limited amount of data available. These plant species don’t have 200 unique stomata. Here overfitting is used, this means that extra information is generated based on the original data. This happens in different ways. In a first phase the original image is mirrored. First over the x-axis, then over the y-axis and then over both axes. Then the image will be rotated 90 degrees and all reflections will repeat themselves. In this way it is possible to go from one image to 8 images. If there are less than 200 stomata, the image will be rotated. The image is rotated in an infinite loop with 25 extra degrees each time. With each rotation a piece of 800 by 600 pixels will be cut out of the image. This until 200 stomata are found.
C. Data preprocessing
Data preprocessing will ensure that the differences between the images are eliminated as much as possible. Data prepro-cessing is used on the training and testing data. However, the data produced for training purposes will differ from the data used for testing. For network testing the data will be normalized. The goal of normalizing data is to make all data as equal as possible. For example, it will ensure that images that are overexposed or underexposed will be adjusted so that the exposure becomes more central. When training a network, data augmentation is done. Here the normalized data will be slightly adjusted to get a robust data set. These minor adjustments ensure that a network can handle a wider spectrum of data.
D. Data normalization
The goal of data normalization is to get each image as consistent as possible. This makes it easier for the neural network to detect stomata. The normalization of data is done
Figure 2: The images with their histogram at the different steps of the normalization process.
with three techniques. A first and simple technique is to convert the RGB color images to grayscale images. This conversion ensures that the differences in color between fresh and dry material as well as the differences between the green and more orange plants are largely reduced.
A second technique uses Adaptive Histogram Equaliztion (AHE). The histogram is a graphical representation of the intensity distribution of an image [2]. With a grayscale image, the histogram will scan all pixels, look at their intensity, and store them cumulatively by their intensity value. AHE is used to improve contrast within an image. This is done by spreading the histogram of an image. The histogram before and after AHE conversion can be seen on the left and middle image in figure 2.
The latter technique uses the gamma transformation. The goal of this transformation is to get the average pixel intensity at 128. This will center the exposure of an image. The gamma transformation therefore mainly affects overexposed and underexposed images. The final image after normalization can be seen in the right part of figure 2.
E. Data augmentation
Data normalisation is used for the test data and aims to get this data as constant as possible. Data augmentation is used on the training data and will make adjustments to the normalized data, making the model more robust.
A first technique will narrow or broaden the histogram. A factor alpha is used to multiply the intensity of each pixel. The factor is randomly chosen between 0.85 and 1.15. After multiplication, a gamma transformation is used to get the average intensity back to 128.
Secondly the image will go through a gamma transforma-tion. A random gamma is choosen between 0.75 and 1.25. A final technique will stretch the image along the x or y axis. A random number is chosen between 1 and 1.2. Next, the x or y axis is stretched by the length of the random number.
III. FASTERR-CNN
A Faster R-CNN is chosen as the basic structure for the neural network. Different structures exist but the Faster R-CNN network is a network that is fast and yet is not just
3
Figure 3: Brief structure of the Faster R-CNN network [4].
designed for the speed [3]. Faster R-CNN is a continuation of the R-CNN network that was developed to recognize objects on images with high accuracy [4]. As the name suggests, Faster R-CNN will be a faster version of the R-CNN network. The Faster R-CNN network (figure 3) consists of three separate models that work together. The first part are ‘the shared layers’, these layers are shared by the next two models. A second model is the Region Proposal Network (RPN). This network transfers the interesting regions to the third model. That third model is responsible for the classification and is called the classifier.
A. Object classification networks
The operation of an object detection network is best ex-plained by using figure 4 and figure 5. Figure 4 shows a VGG16 network. This network works in the same way as the ResNet-50 network but with slightly modified layers. The network is used to classify images. As shown in the figure, an image will enter the network. This image will pass through several layers and after each layer a new feature map will be created. The dimensions of these feature maps become smaller and smaller each time until at the end a 1x1x1000 map remains. So the entire image is shrunk to a 1x1x1000 matrix. Where the 1000 stands for the number of different classes the image can belong to. And the 1x1 stands for the one digit that remains. This is a digit between 0 and 1 that indicates the probability that an image belongs to the given class. This VGG16 network is used to classify an image between 1000 different classes.
The network in figure 4 consists of several layers. Each of these layers is important, but to understand how the feature map can recognize patterns and objects, the convolutional layer is especially important. This layer consists of a series of filters who will slide over the incoming pixels. In the first layers the filters are able to detect straight lines. As more filters are placed one after the other, the filters will be able to detect more complex objects. Figure 5b shows two feature maps of the fifth convolutional layer. The bottom feature map will activate very strongly to angles on an image. The arrow indicates which part of the feature map is activated the most.
Figure 4: Schematic representation of a VGG16 network [5].
Figure 5: Presentation of feature maps. (a) An example image (b) Some feature maps of the conv 5 filters (c) Some other
images that respond strongly to these filters. [6].
The operation of object classification networks should now be clear. In the next paragraph the Faster R-CNN network will be examined more closely. This is an object detection network which makes it slightly different from the object classification network.
B. Object detection network
The object detection network must do the same as the object classification network but on different parts of the figure. With Faster R-CNN these different parts are taken structurally using 9 anchor boxes (figure 6). These anchor boxes are taken at different points in the figure. A stride of 16 is used. This means that the 9 anchor boxes are taken every 16 pixels to insert into the network. All points where the anchor boxes are taken are shown in figure 7. On a 600x800 pixel image with a stride of 16 where 9 anchor boxes are taken at a time, the network will
Figure 6: Square anchors. Figure 7: Grid for anchor boxes.
4
Gewenst True positive TP Detects stomata correctly True negative TN Detects background correctly
Ongewenst False positive FP Detects background as stomata False negative FN Forget to detect a stomata
Table I: Summary of the building blocks for testing a neural network.
generate 16200 anchor boxes. All the anchor boxes go into the shared layer and the feature maps are generated for each anchor box.
The shared layers are used by both the RPN network and the classifier. Because the two networks share the layers, it will not be needed to generate the feature maps twice. Therefore the network will be faster. The shared layers first create feature maps for each of the 16200 anchor boxes. These feature maps all enter the RPN network. The purpose of the RPN network is to distinguish the foreground from the background. This network will not look in which class an box belongs. Of the 16200 anchor boxes the RPN network will forward the best 80 to the classifier. The classifier is now trained to divide the incoming boxes into different classes. In this network the classifier will choose between ‘stomata’ or ‘no stomata’.
IV. METRICS
The Faster R-CNN network consists of three parts, each with its own parameters. In order to set these parameters correctly it is necessary to test the network several times and compare these tests with each other. The comparison of the different tests is mainly based on the F1-score. The F1-score is a combination of the recall and the precision. The parameters rest on four building blocks which are therefore discussed first.
A. Building blocks
The four building blocks are described in table I and are shown in figure 8. The figure contains text for the different boxes. The text indicates the type of building blocks the box represents and the prediction of the model on the box. Also note that there is text for a stomata that is not surrounded by a box. This is because the model did not find this stomata.
B. Precision
Precision is calculated using the following formula. P recision = T P
T P + F P
Precision is the number of correctly detected stomata divided by the number of boxes that the network thinks contain a stomata. So how correct the frames are that are indicated as stomata. The problem with precision is that the network on a 20 stomata image can have a precision of 100% when the model predicts one stomata and nothing else. The model does not make any mistakes in this case but will still miss 19 stomata. The precision doesn’t takes the False Negatives into account.
Figure 8: Visualization of the building blocks.
C. Recall
Recall is calculated using the following formula. Recall = T P
T P + F N
Recall is the number of indicated stomata divided by the total number of stomata present. The recall score will therefore indicate how good the network is in finding all the stomata. The problem with recall is that the network on an image with 20 stomata can have a 100% recall when this model indicates 300 frames as stomata and so happens to indicate the 20 real stomata.The recall does not take the False Positives into account.
D. F1 score
To address the problems of precision and recall, the F1 score is used [7]. The F1 score is calculated using the following formula.
F 1 = 2 ∗ precision ∗ recall precision + recall
By combining precision and recall, the network will be pe-nalized if it indicates too many stomata incorrectly or when it finds too few stomata. Because of this, the F1 score is often used in the validation of neural networks.
V. EXPLORATION OF HYPERPARAMETERS
The Faster R-CNN network consists of several parameters. It is also possible to train on different types of data. During the exploration of the hyperparameters, the optimal parameters are searched to train the network. This is done by adjusting one parameter each time and comparing the results with the previous network. In this part, the optimal hyperparameters are discussed. The hyperparameters have not yet been explained. In order to understand them, background knowledge about Faster R-CNN networks is expected.
After the many tests it appears that the Faster R-CNN network will train optimally on slightly augmented data. The
5
F1 Bij optimale threshold Bij algemene threshold (0,966)
[0 ; 0,5[ 4 16 [0,5 ; 0,8[ 33 38 [0,8 ; 0,85[ 13 76 21 59 [0,85 ; 0,9[ 19 9 [0,9 ; 0,95[ 21 19 [0,95 ; 1] 23 10
Table II: General results of the F1 scores after testing the general model.
techniques and parameters used for light data augmentation are explained in section II-E. The complete data set of 113 plant species contains 10723 images. These images are divided into a part for training and a part for validation. This is done according to a 90-10 split where 90% of the data is used to train the model and 10% is used to validate the model. As test data, 2500 random images are selected from the complete data set. This is from a data set of normalized images. So testing is done on normalized data.
The training of the model is done in two parts. First, the network will train both the RPN model and the classifier. This part will train for 10 epochs. Next the model only trains the classifier for another 10 epochs. At the start of the training an already trained network is loaded in. Because of this it is no longer necessary to train all layers. The first 80 layers are not trained and keep their initial values. The Faster R-CNN network has 143 shared layers. So 80 layers corresponds to 56% of the shared layers.
The learning rate is set to 0.0001. The RPN model will be able to pass a maximum of 80 boxes to the classifier. And the classifier batch size is set to 16. The weight of the unloading functions is set to one.
VI. RESULTS
After the optimal hyperparameters have been examined, the results of the network will be analysed. The examination of the results is divided into a qualitative study and a quantitative study.
A. Quantitative analysis
The F1 scores of the different plant species are plotted in table II. It is assumed that the network can detect stomata correctly when the F1 score is above 0.8. The left part of the table shows the scores when the threshold for each plant species is set separately. There are 76 plant species with a good F1 score. There are also 33 plant species of which the F1 score is not good. But for these 33 plant species the model can still be optimized. There are also four plant species with an F1 score below 0.5. These four plant species have stomata that cannot be detected with a general model. It is possible to train a model specifically for these plant species. But the goal here is to make a global stomata detector.
Within the quantitative research it is possible to investigated whether the size of the stomata has influence the correctness of the detection. In addition, it is also examined whether the oversampling of data causes problems.
Figure 9: Distribution graph of the F1 score in relation to the size of a stomata.
Figure 10: Distribution chart of the F1 score compared to the original number of stomata.
1) Size of stomata: In figure 9 the F1 scores are plotted relative to the size of a stomata. This shows that there is no correlation between the size of a stomata and the F1-score.
2) Oversampelen: Not all plant species have 200 stomata in the original dates. Some species contain more than 200 stomata and others contain only a few stomata. It is possible that the preprocessing technique used, for generating more stomata, does not provide sufficient variation in the data. In figure 10 the number of original stomata are plotted on the F1-score.
From figure 10 can be concluded that the assumption is wrong. It is not the plant species with little original data that are doing badly, but those with a lot of original data. Of the 33 plant species, 15 have more than 200 original stomata. This is 45,45%, while only 14 of the 76 plant species have more than 200 stomata. This corresponds to 18.42%.
B. Qualitative analysis
Qualitative analysis shows that stomata that stand out against their background are better detected. A background that contains little information helps the stomata to stand out from the background (figure 12). Therefore, it is more difficult to detect stomata with a busy background (figure 11). C. Oversampelen
The network currently works well on 76 of the 113 plant species. The aim of the thesis is to develop a network that
6
Figure 11: Dialium pachypyllum, plant species with busy background.
Figure 12: Paramacrolo-bium coeruleum, plant species with flat background.
F1 Optimal threshold Global threshold (0,969)
[0 ; 0,5[ 6 11 [0,5 ; 0,8[ 26 35 [0,8 ; 0,85[ 16 81 22 67 [0,85 ; 0,9[ 27 15 [0,9 ; 0,95[ 22 22 [0,95 ; 1[ 16 8
Table III: General results of the F1 score with extra training on the 33 plant species.
works well on as many plant species as possible. There are different ways to train extra on a part of the data set. Undersampling, oversampling thresholding or cost sensitive learning can be used.
The choice was made to oversample with a factor of two. This means that the 33 plant species will occur twice as much in the training data set. After training with oversampling, the results from table III are obtained. However, the table alone gives a wrong picture. Of the original 33 plant species, 15 now have an F1 score above 0.8. And the F1 score will increase on 25 of the 33 plant species. This with an average of 0.129. However, 10 new plant species have been added that previously had an F1 score greater than 0.8. This can be explained, because of the extra testing on the 33 plant species there is less testing on the plant species with previously a good F1-score. So the F1-score of these plants will decrease slightly.
In general, this new model is better. In a further phase it would be possible to oversample the 10 species of which the F1-score dropped below 0.8. This oversampling will not happen with a factor of two, but rather with a factor of 1.5. It can be further investigated whether this works.
VII. IMPLEMENTATION
The aim of the thesis is to help biologists detect stomata. Biologists should therefore be able to use the developed neural network. To achieve this, a site will be developed and the network will be placed on a server of Ghent University. To secure the servers, the site is placed in a docker container.
The intention is to analyze multiple images at the same time. With this idea in mind the choice has been made to work with zip files. The images that have to be analysed will be put in a .zip file. The zip file is then uploaded to the servers. Here the neural network will analyze the images and put boxes around the stomata. These are then zipped together with a .txt file and
sent back to the user. The .txt file contains all the names of the images that were uploaded combined with their stomata.
VIII. CONCLUSION
In this thesis several neural networks have been investigated that could help biologists detect stomata. Here the choice was made to work with a Faster R-CNN network for the detection. And with a ResNet-50 network as backbone. In order to optimise the use of this network for stomata research, different parameters and metrics were used. These were extensively tested in order to arrive at an ideal combination. The after this the network was tested on the full data set. Here a qualitative and quantitative investigation was used to determine why some stomata were correctly detected and why it was more difficult for the network to detect other stomata. After this investigation one more technique was used to optimise the network.
On the final network 71% of the plants have a F1 score above 0.8. Which means in 71% of the plants the stomata will be detected correctly, or at least good enough to be useful. With further optimization it should be possible to get this number even higher. The final network is then put on the servers. This makes it possible to use this network from all over the world.
REFERENCES
[1] A. M. Hetherington and F. I. Woodward, “The role of stomata in sensing and driving environmental change,” Nature, vol. 424, no. 6951, pp. 901– 908, 2003.
[2] M. Shin, M. Kim, and D.-S. Kwon, “Baseline cnn structure analysis for facial expression recognition,” in 2016 25th IEEE International Symposium on Robot and Human Interactive Communication (RO-MAN), pp. 724–729, IEEE, 2016.
[3] J. Huang, V. Rathod, C. Sun, M. Zhu, A. Korattikara, A. Fathi, I. Fischer, Z. Wojna, Y. Song, S. Guadarrama, et al., “Speed/accuracy trade-offs for modern convolutional object detectors,” in Proceedings of the IEEE conference on computer vision and pattern recognition, pp. 7310–7311, 2017.
[4] S. Ren, K. He, R. Girshick, and J. Sun, “Faster r-cnn: Towards real-time object detection with region proposal networks,” in Advances in neural information processing systems, pp. 91–99, 2015.
[5] K. Simonyan and A. Zisserman, “Very deep convolutional networks for large-scale image recognition,” arXiv preprint arXiv:1409.1556, 2014. [6] K. He, X. Zhang, S. Ren, and J. Sun, “Spatial pyramid pooling in deep
convolutional networks for visual recognition,” IEEE transactions on pattern analysis and machine intelligence, vol. 37, no. 9, pp. 1904–1916, 2015.
[7] T. Saito and M. Rehmsmeier, “The precision-recall plot is more informa-tive than the roc plot when evaluating binary classifiers on imbalanced datasets,” PloS one, vol. 10, no. 3, 2015.
Inhoudsopgave
Lijst van figuren xvii
Lijst van tabellen xxi
1 Inleiding 1
1.1 Belang van huidmondjes . . . 1
1.2 Detectie van stomata met machine learning model . . . 2
2 Data exploratie en preprocessing 6 2.1 Originele data . . . 6
2.2 Genereren van gelijke data . . . 8
2.2.1 Croppen van afbeeldingen . . . 8
2.2.2 Bepalen van het aantal huidmondjes . . . 8
2.3 Normaliseren van de data . . . 9
2.3.1 Histogram equalization . . . 10 2.3.2 Gamma transformatie . . . 15 2.3.3 Totale normalisatie . . . 15 2.4 Data augmentatie . . . 16 2.4.1 Histogram equalization . . . 16 xii
INHOUDSOPGAVE xiii
2.4.2 Gamma transformatie . . . 17
2.4.3 Stretchen van de afbeelding . . . 17
3 Backbone netwerken en werking van machine learning 18 3.1 Onderdelen van backbone netwerken . . . 19
3.1.1 Convolutionele laag . . . 19 3.1.2 Batch normalisatie . . . 20 3.1.3 Activation laag . . . 21 3.1.4 Max pooling . . . 22 3.1.5 Dense laag . . . 23 3.2 VGG16 . . . 23 3.3 ResNet-50 . . . 23
3.3.1 Opbouw ResNet-50 model . . . 26
4 Faster R-CNN 28 4.1 R-CNN . . . 29
4.2 Faster R-CNN . . . 31
4.2.1 De gedeelde lagen . . . 32
4.2.2 Region Proposal Network . . . 33
4.2.3 RPN to RoI . . . 39
4.2.4 Classificatie netwerk . . . 39
5 Parameters en metrieken gebruikt bij model optimalisatie 41 5.1 Verschillende hyperparameters . . . 41
xiv INHOUDSOPGAVE
5.1.2 Aantal te trainen lagen . . . 42
5.1.3 Maximum aantal interessante regio’s . . . 44
5.1.4 Classifier batch grootte . . . 44
5.1.5 Learning rate . . . 44
5.1.6 Gewicht van loss functies . . . 44
5.1.7 Data augmentatie technieken . . . 45
5.2 Vergelijkende metrieken . . . 46
5.2.1 Bouwstenen van de metriek . . . 46
5.2.2 Precisie . . . 47
5.2.3 Recall . . . 48
5.2.4 F1-score . . . 48
5.2.5 Maximum oppervlak onder de precision-recall curve . . . 49
6 Exploratie van hyperparameters 51 6.1 Gebruikte data . . . 51
6.2 Augmentatietechnieken . . . 52
6.2.1 Dieper inzicht op sterk geaugmenteerde data . . . 53
6.3 Trainingsvolgorde . . . 53
6.4 Classifier batch grootte . . . 54
6.5 Learning rate . . . 54
6.6 Aantal te trainen lagen . . . 55
6.7 Maximum aantal interessante kaders . . . 56
6.7.1 Maximum aantal interessante kaders tijdens de training . . . 56
INHOUDSOPGAVE xv
6.8 Gewichten van loss functies . . . 58
6.9 Testen met de volledige dataset . . . 59
6.10 Vergelijking aantal cycli . . . 60
6.11 Trainen op niet-geaugmenteerde data . . . 61
6.12 Conclusie . . . 62
7 Discussie 64 7.1 Kwantitatief onderzoek . . . 64
7.1.1 De grootte van de huidmondjes . . . 65
7.1.2 Fout bij oversampeling . . . 67
7.1.3 Concentratie huidmondjes per afbeelding . . . 68
7.2 Kwalitatief onderzoek . . . 68
7.2.1 Sterke achtergrond . . . 68
7.2.2 Foute labeling . . . 70
7.3 Oversampelen . . . 70
7.3.1 Resultaten . . . 72
7.4 Testen op nieuwe data . . . 75
7.4.1 Huidmondjes die correct gedetecteerd zijn . . . 76
7.4.2 Huidmondjes die middel gedetecteerd zijn . . . 78
7.4.3 Huidmondjes die slecht gedetecteerd zijn . . . 78
7.4.4 Huidmondjes die niet handmatig gevonden worden . . . 79
7.5 Conclusie . . . 79
8 Implementatie 92 8.1 Gebruiken van het neurale netwerk . . . 92
xvi INHOUDSOPGAVE 8.1.1 Uploaden van zip bestanden . . . 93 8.2 Opbouw achterliggende code . . . 94
9 Conclusie 96
Lijst van figuren
1.1 Voorbeelden van stomata op enkele verschillende plantsoorten. . . 3
1.2 Globaal overzicht van een classificatie model. . . 4
1.3 Voorstelling van feature maps (a) Een voorbeeld afbeelding (b) Enkele feature maps van de conv5 filters (c) Enkele andere afbeeldingen die sterk reageren op deze filters. [1]. . . 5
2.1 Input data: Blad met bounding boxen. . . 7
2.2 Voorbeeld van een stuk van de input data. . . 7
2.3 Bekijken van het histogram voor en na histogram equalization. . . 11
2.4 Bekijken van het histogram voor en na adaptive histogram equalization. . . 11
2.5 Clippen van het histogram bij CLAHE. . . 12
2.6 Vergelijking van verschillende clip limieten met een tile grootte van (32,32) bij Contrast Limited Adaptive Histogram Equalization. . . 13
2.7 Vergelijking van verschillende tile groottes met een clip limiet van 1,4 bij Contrast Limited Adaptive Histogram Equalization. . . 14
2.8 De originele figuur vergeleken met de figuur na CLAHE en de figuur normalisatie. 15 3.1 Voorbeeld van de werking van een convolutionele laag op een 2D afbeelding . . . 19
3.2 Filter om verticale lijnen mee te detecteren . . . 20
3.3 Filter om horizontale lijnen mee te detecteren . . . 20 xvii
xviii LIJST VAN FIGUREN
3.4 De sigmoid en ReLU activatiefuncties . . . 22
3.5 Max pooling laag met een stride van 2 en een window van 2 [2] . . . 23
3.6 De architectuur van een VGG16 netwerk [3] . . . 24
3.7 Training loss (links) en test loss (rechts) op CIFAR-10 met 20-lagen en 56-lagen [4] 25 3.8 Resudal learning blok [4] . . . 25
3.9 Het loss oppervlak van een ResNet-56 netwerk met en zonder skip connecties [5]. 26 3.10 Identity blok . . . 27
3.11 Convolutionele blok . . . 27
4.1 Accuratie van de verschillende modellen ten opzichte van de GPU tijd [6]. . . 29
4.2 Beknopte opbouw van R-CNN [7]. . . 30
4.3 Beknopte opbouw van Faster R-CNN [8]. . . 31
4.4 De verandering van de dimensies van een input afbeelding door de verschillende gedeelde lagen. . . 33
4.5 Voorstelling van een rooster op de input afbeelding. . . 34
4.6 Afbeelding met één set anchors op afgebeeld. . . 35
4.7 Formule voor het berekenen van Intersection over Union (IoU). . . 36
4.8 Enkele voorbeelden van de waardes verbonden aan Intersection over Union (IoU). 36 4.9 Plots van de L1, L2 en smooth L1 loss functie. Dit met δ = 1. . . 38
4.10 Plots van de cross entropy loss functie. . . 38
5.1 De lagen van het gemeenschappelijke netwerk. . . 43
5.2 Afbeelding met een grote precisie. . . 48
5.3 Afbeelding met een grootte recall. . . 48
LIJST VAN FIGUREN xix 6.1 Weergave van kaders die van het RPN model naar de classifier gaan. Dit bij de
Ipomoea cairica De Wild plantsoort. . . 57
6.2 Test van een netwerk getraind op niet genormaliseerde data. . . 62
6.3 Test van een netwerk getraind op genormaliseerde data. . . 62
7.1 De vier plantsoorten met een F1-score lager dan 0,5. . . 66
7.2 Spreidingsgrafiek van de F1-score ten opzichte van de grootte van een huidmondje. 67 7.3 Spreidingsgrafiek van de F1-score ten opzichte van het oorspronkelijk aantal huid-mondje. . . 69
7.4 Het gemiddeld aantal huidmondjes per soort wordt vergeleken met het gemiddeld aantal huidmondjes per afbeelding. . . 69
7.5 Alpinia sanderaes met F1-score = 0,5102. . . 70
7.6 Brassia met F1-score = 0,8467. . . 70
7.7 De verandering in F1-score op het verschil van 200 met de grootte van een huid-mondje in absolute waarde. . . 73
7.8 Enkele plantsoorten met een F1-score die is verlaagd terwijl er extra data wordt toegevoegd. . . 74
7.9 Huidmondje is in twee gesneden. . . 76
7.10 Voorbeelden van huidmondjes die het netwerk goed herkent. . . 77
7.11 Enkele plantsoorten waarop het netwerk de huidmondjes goed herkent. . . 77
7.12 Enkele plantsoorten waarop het netwerk de huidmondjes gemiddeld herkent. . . . 78
7.13 Enkele plantsoorten waarop het netwerk de huidmondjes gemiddeld herkent. . . . 79
7.14 Afbeeldingen waarop het netwerk goed de huidmondjes kan detecteren. . . 81
7.15 Afbeeldingen waarop het netwerk goed de huidmondjes kan detecteren. . . 82
7.16 Afbeeldingen waarop het netwerk goed de huidmondjes kan detecteren. . . 83
xx LIJST VAN FIGUREN 7.18 Afbeeldingen waarop het netwerk goed de huidmondjes kan detecteren. . . 85 7.19 Afbeeldingen waarop het netwerk gemiddeld de huidmondjes kan detecteren. . . 86 7.20 Afbeeldingen waarop het netwerk gemiddeld de huidmondjes kan detecteren. . . 87 7.21 Afbeeldingen waarop het netwerk gemiddeld de huidmondjes kan detecteren. . . 88 7.22 Afbeeldingen waarop het netwerk slecht de huidmondjes kan detecteren. . . 89 7.23 Afbeeldingen waarop het netwerk slecht de huidmondjes kan detecteren. . . 90 7.24 Afbeeldingen waarvan het niet duidelijk is wat de huidmondjes zijn . . . 91 8.1 Site wanneer deze juist wordt gestart. . . 93 8.2 Uploaden van een zip bestand. . . 93 8.3 Een zip bestand is gekozen. . . 93 8.4 Site is aan het laden. . . 94 8.5 Downloaden van een zip bestand. . . 94 8.6 Achterliggende werking van de site. . . 95 8.7 De mappen voor of na het runnen van de code. . . 95 8.8 De mappen wanneer ‘de_zip.zip’ wordt geüpload. . . 95
Lijst van tabellen
5.1 Samenvatting van de verschillende augmentatie technieken. . . 46 5.2 Samenvatting van de bouwstenen voor het testen van een neuraal netwerk. . . 47 6.1 Resultaten van de netwerken getraind met verschillend geaugmenteerde data. En
getest op de validatie set die bestaat uit genormaliseerde data. . . 52 6.2 Resultaten van een netwerk getraind op sterk geaugmenteerde data en getest op
de de validatie data, licht geaugmenteerde data en sterk geaugmenteerde data. . 53 6.3 Vergelijking training in één stuk met training in twee stukken. Bij het model dat
in één stuk is getraind zijn RPN en classifier constant getraind. Bij het model dat in twee stukken is getraind zijn eerst het RPN en classifier model getraind en vervolgens allen de classifier. . . 54 6.4 Vergelijking van de classifier batch grootte op data met licht augmentie. . . 54 6.5 Vergelijking van de learning rate op licht augmente data. . . 55 6.6 Vergelijking van de verschillende te trainen blokken. Alle lagen t.e.m. de vermelde
blok behouden hun initiële waarde . . . 56 6.7 Vergelijking van het maximum aantal kaders bij de training van een model. . . . 57 6.8 Het maximum aantal kaders tijdens de test fase. . . 58 6.9 Test tijd met een verschillend aantal maximum kaders. . . 58 6.10 Resultaten van verschillende λ waardes bij een Faster R-CNN netwerk [8]. . . 59 6.11 Test met verschillende gewichten voor de loss functies . . . 59 6.12 Vergelijking netwerk met 60 cycli en netwerk met 200 cycli. . . 60
xxii LIJST VAN TABELLEN 6.13 Vergelijking verschillende soorten data op de hele dataset. . . 61 6.14 Samenvattende tabel met de uiteindelijke parameters en data op de korte dataset. 63 7.1 Algemene resultaten van de F1-scores na het testen van het algemene model. . . 65 7.2 De vier plantsoorten met een F1-score lager dan 0,5 in het originele netwerk . . . 66 7.3 Lijst van plantsoorten met een F1-score tussen 0,5 en 0,8. . . 71 7.4 Algemene resultaten van de F1-score bij extra training op de 33 plantsoorten. . . 75
Lijst van afkortingen en symbolen
R-CNN Region-based Convolutional Neural Network
Faster R-CNN Faster Region-based Convolutional Neural Network YOLO You Only Look Once
SSD Single Shot Detector RPN Region Proposal Network IoU Intersection over Union MAE Mean Average Error MSE Mean Square Error RoI Region of Interest
NMS Non-maximum Suppression VGG16 Visual Geometry Group CNN Convolutional Neural Network ReLU Rectified Linear Unit
mAP mean Average Precision
R-FCN Region-based Fully Convolutional Networks CO2 koolstofdioxides
1
Inleiding
1.1 Belang van huidmondjes
Planten zijn in staat om koolstofverbindingen op te bouwen. Deze plantaardige organismen hebben koolstofverbindingen nodig om te overleven. Om de koolstofverbindingen op te bouwen hebben planten water, koolstofdioxide (CO2) en licht nodig. De opname van CO2, nodig voor dit proces, gebeurt vooral via de bladeren. Om uitdroging en ziektes tegen te gaan zijn deze bladeren echter beschermd met een waslaag. Door deze waslaag zal het blad echter geen CO2 kunnen opnemen. Om deze opname van CO2 toch mogelijk te maken zijn er huidmondjes aanwezig op het blad. Deze huidmondjes zijn vergelijkbaar met poriën bij de mens. Wanneer de huidmondjes open staan zal het mogelijk zijn voor de plant om gassen (CO2, waterdamp en O2) uit te wisselen met de atmosfeer. Wanneer het warm wordt zal de plant zijn huidmondjes sluiten, dit om uitdroging tegen te gaan [9].
De vorming van huidmondjes is afhankelijk van omgevingsfactoren. Hierdoor is het mogelijk om uit de grootte en dichtheid van deze huidmondjes het klimaat te reconstrueren waarin deze plant leefde. Onder andere de temperatuur, atmosfeer en CO2-concentratie zijn hieruit af te leiden [10]. Biologen zullen deze eigenschappen bijgevolg gebruiken om onder andere de verandering van het klimaat in kaart te brengen [11].
2 HOOFDSTUK 1. INLEIDING De omstandigheden waarin het blad is geproduceerd zullen ook een invloed hebben op de grootte en dichtheid van stomata (huidmondjes). Dit kunnen locale veranderingen zijn, zoals bladeren die gevormd zijn in een koude en natte winter zijn groot en bevatten meer stomata . Dit terwijl de bladeren geproduceerd in een droge zomer eerder klein zijn en minder stomata bevatten [12]. Maar ook verschillen op lange termijn spelen een rol. Zoals de CO2-concentratie in de atmosfeer. Wanneer er veel CO2in de atmosfeer zit kunnen planten met minder stomata overleven. Wanneer de hoeveelheid CO2 in de atmosfeer daalt zullen planten met meer stomata in het voordeel zijn en dus een hogere overlevingskans hebben [12]. Door de evolutie van de grootte van stomata op te volgen kunnen biologen de CO2-concentratie doorheen de jaren reconstrueren. Zo wordt de 400 miljoen jaar tijdens het Fanerozoïcum tijdperk, geassocieerd met een lage CO2-concentratie, gekenmerkt door bladeren met een hoge concentratie aan stomata.
Door het belang van huidmondjes in planten wordt er veel onderzoek gedaan op deze microsco-pische poriën. Tijdens een onderzoek is het vaak belangrijk om de grootte en concentratie van deze stomata te achterhalen. Wanneer meerdere planten uit verschillende locaties met elkaar vergeleken worden, is het vaak een hele taak om al deze stomata in kaart te brengen. Momenteel tellen onderzoekers deze stomata handmatig en slaan ze de eigenschappen ervan op. In figuur 1.1 zijn er drie plantsoorten afgebeeld elk met hun eigen stomata. Het is duidelijk dat het een duur en tijdrovend proces is om de stomata handmatig aan te duiden. Het zou dus een enorme hulp zijn moest er een programma bestaan dat deze stomata automatisch aanduidt.
1.2 Detectie van stomata met machine learning model
Door de grote vooruitgang in machine learning modellen is het vandaag de dag mogelijk om een programma te ontwikkelen dat stomata automatisch detecteert. Het detecteren van stomata kan ook via de klassieke beeldverwerking. Met klassieke beeldverwerking is het echter moeilijk om een model te maken dat op de verschillende plantsoorten goed zal werken. Het is hier wel de bedoeling dat één model in staat is om huidmondjes te herkennen op verschillende plantsoorten. Een neuraal netwerk is hiertoe in staat en daarom wordt er verder ook met een neuraal netwerk gewerkt. Om de verdere masterproef goed te kunnen volgen wordt hier al een kort overzicht gegeven van hoe een neuraal netwerk in staat is om objecten te detecteren. Voor deze uitleg wordt er gebruik gemaakt van een classificatie netwerk. Een classificatie netwerk heeft als doel afbeeldingen te classificeren. Er wordt dus telkens naar de volledige afbeelding gekeken.
In figuur 1.2 wordt de structuur van een classificatie model afgebeeld. Er is te zien hoe de afbeelding die geclassificeerd moet worden door verschillende lagen gaat. Na de eerste laag wordt er niet meer over een afbeelding gesproken maar over een feature map. Dit is een map die aanduidt waar de features zich bevinden. Na de eerste laag zijn deze features zeer eenvoudig. Het model zal dan in staat zijn om rechte lijnen te herkennen op afbeeldingen. Na meerdere lagen
1.2. DETECTIE VAN STOMATA MET MACHINE LEARNING MODEL 3
(a) Styrchnos usambarensis (b) Trilepisium madagascariense.
(c) Ipomaea eriocarpa Faulkner.
4 HOOFDSTUK 1. INLEIDING
Figuur 1.2: Globaal overzicht van een classificatie model.
is het netwerk in staat om iets complexere features te detecteren. Enkele voorbeelden hiervan zijn: hoeken, cirkels of ovalen. En na nog wat lagen is het netwerk in staat om zeer complexe objecten te detecteren zoals vogels, boten of huidmondjes.
Het classificatie netwerk bestaat uit verschillende lagen die elke van belang zijn. Om de werking van het neurale netwerk te begrijpen is het vooral belangrijk om de convolutionele laag te verstaan. Deze laag bestaat uit een reeks filters die over de binnenkomende pixels gaan. In de eerste lagen zijn de filters in staat om rechte lijnen te detecteren. In figuur 1.2 zijn de dimensies van de afbeelding en feature maps telkens weergegeven. In het begin is dit een 224x224x3 kleur afbeelding. Er zijn dus 3 lagen in de diepte (z-as) voor RGB. Na de eerste laag zijn de dimensies 224x224x64, er zijn dus 64 feature maps gecreëerd van de afbeelding. Door verschillende feature maps te combineren is het mogelijk om complexere objecten te detecteren. Zo kan een feature map die horizontale lijnen detecteert gecombineerd worden met een feature map die verticale lijnen detecteert. De combinatie van deze twee feature maps zal hoeken detecteren.
In figuur 1.3b zijn twee feature maps te zien van de vijfde convolutionele laag. De onderste van deze filters zal zeer sterk reageren op hoeken in een afbeelding. Met de pijl is aangeduid welk deel van de feature map er het meest geactiveerd wordt.
De uiteindelijke dimensie van het classificatie netwerk in figuur 1.2 is 1x1x1000. De 1000 in de laatste laag duidt aan hoeveel verschillende klassen er zijn. Elke klasse krijgt nog een 1x1 matrix. Deze matrix bevat één cijfer tussen 0 en 1 dat aangeeft hoeveel kans er is dat de originele afbeelding tot een bepaalde klasse behoort.
1.2. DETECTIE VAN STOMATA MET MACHINE LEARNING MODEL 5
Figuur 1.3: Voorstelling van feature maps (a) Een voorbeeld afbeelding (b) Enkele feature maps van de conv5 filters (c) Enkele andere afbeeldingen die sterk reageren op deze filters. [1]. Een classificatie model kijkt enkel naar de volledige afbeelding terwijl een detectie model ook naar specifieke delen binnen een afbeelding kijkt. Om huidmondjes te detecteren wordt er dus met een detectie model gewerkt. Dit detectie model is iets ingewikkelder en hoe dit precies zit wordt verder in hoofdstuk 3 en hoofdstuk 4 van deze masterproef besproken. Het is duidelijk dat de data waarop een neuraal netwerk getraind wordt een zeer grote impact heeft. Het is mo-gelijk om de data die wordt gebruikt te bewerken. In het hoofdstuk 2 wordt de data besproken. Er wordt onderzocht welke data er beschikbaar is en hoe deze bewerkt wordt voor het trainen en testen van het neurale netwerk. Verder worden de parameters en metrieken besproken die gebruikt worden om het model te trainen en te testen. In hoofdstuk 6 worden de verschillen-de parameters geanalyseerd. En wordt gezocht naar verschillen-de optimale waarverschillen-de voor elke parameter. Uiteindelijk worden de resultaten besproken in hoofdstuk 7. Er wordt ook gekeken naar hoe goed het uiteindelijke model is en op welke data het model beter of slechter werkt. In hoofdstuk 8 wordt gekeken hoe het model geïmplementeerd wordt. Hoe en waar onderzoekers het model kunnen gebruiken. In hoofdstuk 9 wordt alles nog eens kort samengevat en wordt er gekeken wat er nog kan gebeuren om het model te verbeteren.
Het uiteindelijke doel is om een model te creëren dat robuust werkt op de verschillende plant-soorten. Het moet een model zijn dat door verschillende onderzoekers gebruikt kan worden die elk onderzoek doen op verschillende plantsoorten. Om dit alles mogelijk te maken wordt er ook een site ontwikkeld die zal draaien op de servers van Universiteit Gent. Zo zullen onderzoekers van over de hele wereld dit model kunnen gebruiken.
2
Data exploratie en preprocessing
Data preporcessing is een van de belangrijkste onderdelen in een neuraal netwerk. Zonder de correcte data is een dergelijk model niet in staat om te leren. Het is dus belangrijk dat de data in een juiste vorm staat en dat deze correct gelabeld is. Omdat er hier is gewerkt met 113 verschillende plantsoorten opgedeeld, in meer dan 900 afbeeldingen, zal het belangrijk zijn om dit process volledig te optimaliseren. In dit hoofdstuk zal er dus gekeken worden hoe deze optimalisatie is gebeurd en waar er belangrijke beslissingen zijn genomen.
2.1 Originele data
Input data is een van de belangrijkste onderdelen voor het trainen van een neuraal netwerk. Deze data bestaat uit afbeeldingen gecombineerd met een .txt bestand waarin de bounding boxen per afbeelding worden meegegeven. In figuur 2.1 wordt een afbeelding weergegeven waarbij de bounding boxen zijn afgebeeld. Natuurlijk zullen deze niet worden afgebeeld wanneer het model getraind zal worden.
Deze figuren werden reeds allemaal gelabeled voordat ze in dit onderzoek terecht kwamen. Het label.txt document waarin de labels stonden, bevatte voor elke afbeelding een lijst van al zijn huidmondjes. Een verkorte vorm van deze lijst is weergegeven in figuur 2.2 en bevatte voor elk
2.1. ORIGINELE DATA 7
Figuur 2.1: Input data: Blad met bounding boxen. huidmondje de coördinaten van het centrum.
Voor het trainen van een model dat de bounding boxen moet achterhalen is het dus nodig om deze bounding boxen ook mee te geven. Hiervoor is een stuk code geschreven dat alle afbeeldingen overloopt en waarin vervolgens manueel de grootte van de bounding boxen wordt meegegeven. Er werd vervolgens gekeken of het centrum wel goed bepaald was en wanneer dit niet het geval was werd dit centrum aangepast. Het is namelijk enorm belangrijk dat de input data correct gelabeld is. Wanneer er zich fouten voordoen in deze data zal het neuraal netwerk deze fouten proberen te verwerken. Hierdoor zal dit model niet meer correct werken. Daarom is het belangrijk om de data volledig op te kuisen.
labels.txt x;y;Foto;
1120.4893;192.0489;Strychnos spinosa_Berhaut 131_blad1-vak1.jpg; 948.0122;293.5780;Strychnos spinosa_Berhaut 131_blad1-vak1.jpg; 1130.2753;628.7462;Strychnos spinosa_Berhaut 131_blad1-vak1.jpg; 1385.9327;755.9633;Strychnos spinosa_Berhaut 131_blad1-vak1.jpg; 1289.2966;1000.6116;Strychnos spinosa_Berhaut 131_blad1-vak1.jpg; 1542.5077;1147.4006;Strychnos spinosa_Berhaut 131_blad1-vak1.jpg;
8 HOOFDSTUK 2. DATA EXPLORATIE EN PREPROCESSING
2.2 Genereren van gelijke data
Zoals reeds aangehaald is de input data enorm belangrijk. Het is vooral belangrijk dat de data constant is. Dit betekent dat alle afbeeldingen even groot zullen zijn en dat elke plantsoort een gelijk aantal huidmondjes bevat.
2.2.1 Croppen van afbeeldingen
Wanneer naar de eigenschappen van de data wordt gekeken, is er te zien dat dit bestaat uit een reeks RGB afbeeldingen met een dimensie van 1200 op 1600 pixels. Het is mogelijk om dit rechtstreeks in te geven als input voor het neuraal netwerk. Maar wanneer er getraind wordt op zo grote afbeeldingen zal dit zeer traag verlopen. Dit is dus niet aan te raden.
Het is dus beter om kleinere afbeeldingen als input mee te geven. Er zijn twee manieren om dit te bekomen. Ofwel kan de afbeelding geschaald worden ofwel kan deze uitgesneden worden. Bij het schalen van een afbeelding zal er echter data verloren gaan, dus dit is geen ideale oplossing. Het gebruikte model zal de afbeelding ook schalen waardoor deze nog kleiner wordt. Hierdoor is de keuze gemaakt om de afbeeldingen in stukken op te delen. Aangezien het model goed werkt op een afbeelding met een korte zijde van 600 pixels is er gekozen om de originele afbeelding in 4 te splitsen. Zo ontstaan er 4 delen van elk 600 op 800 pixels.
2.2.2 Bepalen van het aantal huidmondjes
Een volgend belangrijk aspect is dat er van elke plantensoort ongeveer evenveel huidmondjes te vinden zijn. Dit omdat een model anders kan overfitten op bepaalde plantsoorten terwijl andere soorten nauwelijks herkend worden. Uit voorgaand onderzoek bleek dat 200 huidmondjes per plantensoort voldoende zijn. Het is dus belangrijk om alle plantsoorten te overlopen en na te gaan hoeveel huidmondjes deze bevatten. Wanneer dit er meer dan 200 zijn zullen afbeeldingen verwijderd worden en wanneer dit er minder dan 200 zijn zullen enkele technieken worden toegepast om extra data te creëren. Hiervoor is dus een stuk code geschreven dat volgende onderdelen bevat.
De code zal in een eerste stap kijken hoeveel huidmondjes de plantsoort bevat. Vervolgens zullen alle afbeeldingen van de bepaalde plantsoort in 4 worden gesneden en zullen deze worden opgeslagen. Wanneer de kaap van 200 huidmondjes wordt bereikt zal gestopt worden met het opslaan van huidmondjes. Wanneer deze niet wordt bereikt en alle afbeeldingen reeds overlopen zijn zal er worden overgegaan naar een volgende manier om data te genereren. Dit zal in een eerste stap gebeuren door de afbeelding te spiegelen over de horizontale as. Na het spiegelen zal
2.3. NORMALISEREN VAN DE DATA 9 de afbeelding weer in 4 worden gesneden en opgeslagen. Als er nu nog niet genoeg huidmondjes zijn dan zal de afbeelding gespiegeld worden over de verticale as. Vervolgens kan de afbeelding gespiegeld worden over zowel de horizontale als de verticale as. En in een laatste stap kan de afbeelding 90 graden gedraaid worden en kunnen voorgaande technieken zich herhalen.
Door deze 8 eenvoudige stappen is het mogelijk om van een plantensoort met 25 huidmondjes over te gaan tot een reeks afbeeldingen voor deze plantsoort met een totaal van 200 huidmondjes. Het probleem is hier echter dat sommige plantsoorten minder dan 25 huidmondjes bevatten. Het zal dus nodig zijn om nog een laatste techniek toe te passen om ook voor deze soorten voldoende data te bekomen. Hier is er gekozen om de afbeelding telkens 25 graden te roteren en om uit de geroteerde afbeelding een stuk te snijden van 600 op 800 pixels. Dit kan vervolgens in een oneindige loop worden gestoken om zo zelf van één huidmondje naar 200 huidmondjes over te gaan. Er is gekozen voor 25 graden omdat dit niet deelbaar is door 360. Zo is het mogelijk de afbeelding tot 72 keer te draaien voordat er in herhaling wordt gevallen. Wanneer er na 72 keer nog steeds geen 200 huidmondjes zijn gevonden zal de loop toch verder doorgaan tot er 200 huidmondjes zijn gevonden. Hier is nog als extra toegevoegd dat er na 15 keer gespiegeld wordt over de verticale as en na 30 keer zal er gespiegeld worden over de horizontale as. Zo zal er worden verder gewerkt met een gespiegelde afbeelding.
Nu het mogelijk is om van elke plantsoort 200 unieke huidmondjes te genereren kan er over gegaan worden naar het normaliseren van de data.
2.3 Normaliseren van de data
Het normaliseren van de input data is een volgende belangrijke stap. Het is belangrijk dat de input data altijd onder een gelijke vorm wordt aangebracht. Het is namelijk mogelijk dat afbeeldingen over- of onderbelicht zijn. Of dat de focus wat fout zit en de afbeelding hierdoor zeer scherp of juist zeer wazig wordt. Wanneer een neuraal netwerk is getraind op data die steeds overbelicht was maar met een goede focus dan zal dit model de data goed kunnen herkennen. Wanneer er vervolgens een onderbelichte of on-gefocuste afbeelding toekomt dan zal dit model hier last mee hebben en de huidmondjes mogelijks niet herkennen.
In een eerste stap zullen de RGB afbeeldingen worden omgezet naar grijswaarde afbeeldingen. Dit is een van de meest eenvoudige manieren van data normalisatie. Het is nuttig om dit te doen omdat sommige input afbeeldingen zijn genomen op verse bladeren terwijl andere zijn genomen op gedroogde bladeren. De verse bladeren hebben de neiging om iets groener te zijn dan de gedroogde waarde. Wanneer alle afbeeldingen naar grijswaarde worden gezet zullen deze verschillen ongedaan gemaakt worden. Er zijn ook enkele bladeren iets roder getint dan de andere bladeren. Ook deze verschillen worden vermeden wanneer er wordt overgegaan naar grijswaarde.
10 HOOFDSTUK 2. DATA EXPLORATIE EN PREPROCESSING
2.3.1 Histogram equalization
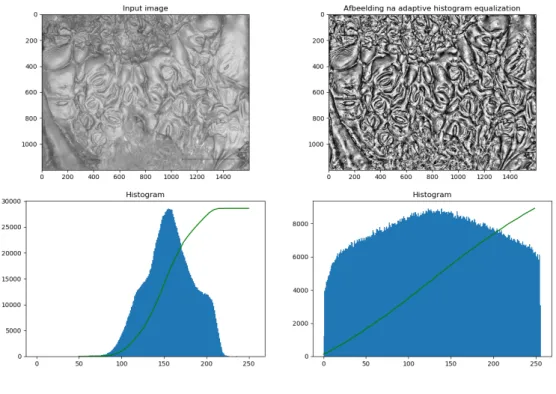
Na het bekijken van enkele onderzoeken rond normalisatie technieken bleek dat histogram equa-lization een van de meest geschikte normalisatie technieken is bij machine learning [13]. Het histogram is een grafische voorstelling van de intensiteit distributie van een afbeelding. Bij een grijswaarde afbeelding zal het histogram alle pixels overlopen, hun intensiteit bekijken en deze cumulatief per intensiteit waarde opslaan in een grafiek. Histogram Equalization (HE) is een techniek die wordt gebruikt om het contrast binnen een afbeelding te verbeteren. Dit wordt gedaan door het histogram van de afbeelding uit te spreiden. Er zijn meerdere technieken om dit te bekomen maar hier is er gekozen voor Adaptive Histogram Equalization (AHE). Bij deze techniek zal er telkens naar een deel van de afbeelding worden gekeken en zal het histogram binnen dit deel genormaliseerd worden. Het voordeel van adaptive histogram equalization is dat er geen volledige vlakken over- of onderbelicht zullen worden. Adaptive histogram equalization heeft als nadeel dat het volledige spectrum niet optimaal gebruikt zal worden.
In figuur 2.3 wordt de input figuur meegegeven alsook de figuur na histogram equalization. Bij elk van de figuren wordt ook het histogram en het cumulatief histogram meegegeven. Het is duidelijk te zien dat het cumulatief histogram een bijna lineaire rechte wordt na de transformatie van de afbeelding. Dit betekent dat de intensiteit mooi verspreid is over de hele figuur. Het valt wel op dat de afbeelding zeer lichte en donkere stukken bevat na het uitvoeren van histogram equalization. In deze stukken van de afbeelding zal er informatie verloren gaan.
Deze donkere en lichte vlakken ontstaan omdat er bij histogram equalization direct naar de volledige afbeelding wordt gekeken. Het is mogelijk om dit fenomeen te vermijden door telkens naar kleinere stukken van de afbeelding te kijken. Dit wordt adaptive histogram equalization genoemd. Bij deze techniek zal de figuur worden opgedeeld in verschillende blokken genaamd ’tiles’. Vervolgens zal op elke blok histogram equalization worden toegepast. Hierdoor zullen donkere of lichtere delen van de afbeelding vergeleken worden met de omliggende pixels die bijgevolg ook donkerder of lichter zijn dan de algemene afbeelding. Dit heeft als gevolg dat deze delen van de figuur niet volledig donker of licht worden gezet maar genormaliseerd zullen worden ten opzichte van de omliggende pixels. Hierdoor zullen de plaatselijke contrasten vergroot worden. In figuur 2.4 is de input afbeelding te zien met daarnaast de afbeelding na adaptive histogram equalization.
Het is in de meeste gevallen een betere oplossing omdat nu de lokale contrasten versterkt worden en er geen informatie verloren zal gaan. Adaptive histogram equalization kan in sommige gevallen ook voor problemen zorgen. Dit wanneer een lokaal deel van de figuur veel ruis bevat of wanneer een lokaal deel van de figuur bijna geen contrast bevat. In het eerste geval zal de ruis versterkt worden wat nooit de bedoeling is. In het tweede geval zal AHE ervoor zorgen dat er toch contrasten ontstaan. Hierdoor zal deze functie van een regio met weinig tot geen contrast ook
2.3. NORMALISEREN VAN DE DATA 11
Figuur 2.3: Bekijken van het histogram voor en na histogram equalization.
12 HOOFDSTUK 2. DATA EXPLORATIE EN PREPROCESSING
Figuur 2.5: Clippen van het histogram bij CLAHE.
een regio maken met veel contrast. Dit kan voor problemen zorgen. Er bestaat gelukkig een functie genaamd ’Contrast Limited Adaptive Histogram Equalization’ (CLAHE) die het tweede probleem zal voorkomen. Hier zal het versterken van contrasten gelimiteerd worden zodat er geen contrasten ontstaan waar deze er origineel niet waren.
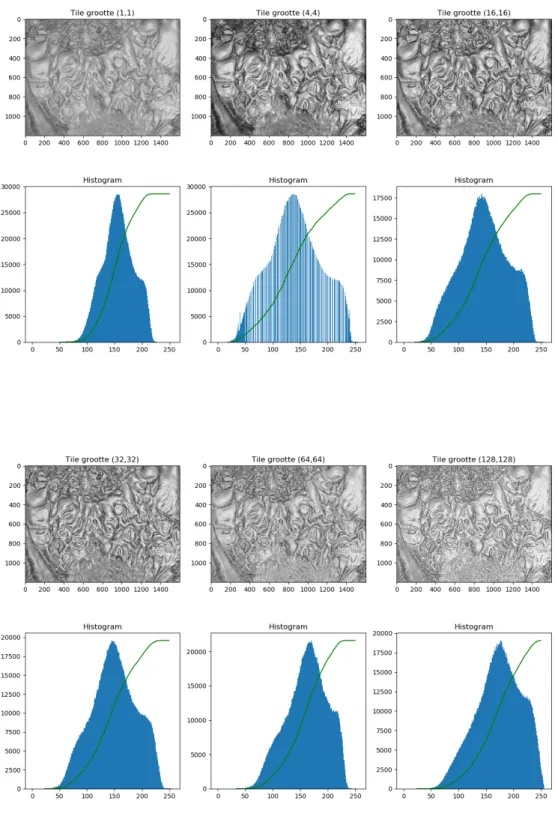
CLAHE zal dit vermijden door een clip te zetten op een bepaalde hoogte in het histogram [14]. Vervolgens zullen alle waarden boven deze clip worden afgesneden en terug verdeeld naar het histogram. Dit is te zien in figuur 2.5. Dankzij deze clip zal het histogram uitgesmeerd worden en zo zullen grote locale versterkingen vermeden worden.
Nu geweten is hoe de ruis en uitschieters vermeden kunnen worden, is het tijd om verschillende waarden uit te testen. CLAHE zal hetzelfde reageren als de normale histogram equalization wanneer de clip groot is en de tile size gelijk aan 1 wordt gezet. Wanneer de clip zeer laag wordt gezet, tot waarden rond de 0,001, en de tile size onder de (4,4) blijft dan zal CLAHE maar weinig invloed hebben op het histogram en op de figuur. Met deze informatie wordt er gezocht naar een optimale clip en tile size om de figuren te normaliseren.
Er worden enkele testen uitgevoerd waar de clip en tile size eerst afzonderlijk worden aangepast en vervolgens worden gecombineerd. In figuur 2.6 worden verschillende clip limieten vergeleken. Dit bij een tile size van telkens (32,32). De originele afbeelding is te zien in figuur 2.3 en werd hier niet herhaald. Er is te zien dat een kleine clip limiet het histogram en bijgevolg de figuur weinig zal beïnvloeden en alleen grotere lokale contrasten zal uitvergroten. Terwijl een grote clip limiet ervoor zal zorgen dat er zelfs contrasten ontstaan in vlakke stukken.
In figuur 2.7 worden verschillende tile groottes getest. Hier is er telkens een clip waarde van 1,4 gekozen. Hieruit bleek dat een tile grootte van (32,32) gecombineerd met een clip van 1,4 optimaal leek voor het normaliseren van de data. Hier kan later op terug gekomen worden indien uit verdere testen zou blijken dat dit geen goede normalisatie is voor het machine learning netwerk.
2.3. NORMALISEREN VAN DE DATA 13
Figuur 2.6: Vergelijking van verschillende clip limieten met een tile grootte van (32,32) bij Contrast Limited Adaptive Histogram Equalization.
14 HOOFDSTUK 2. DATA EXPLORATIE EN PREPROCESSING
Figuur 2.7: Vergelijking van verschillende tile groottes met een clip limiet van 1,4 bij Contrast Limited Adaptive Histogram Equalization.
2.3. NORMALISEREN VAN DE DATA 15
Figuur 2.8: De originele figuur vergeleken met de figuur na CLAHE en de figuur normalisatie.
2.3.2 Gamma transformatie
Een laatste normalisatie techniek is het centreren van het histogram. Deze techniek heeft als doel om de gemiddelde intensiteit rond de 128 te krijgen, dit omdat 128 de helft is van 255 wat het verschil is tussen de maximale en minimale intensiteit. Zo zal vermeden worden dat er over- of onderbelichte afbeeldingen overblijven in de genormaliseerde data. Het centreren van het histogram zal worden bereikt met een gamma transformatie. Er zal naar elke afbeelding afzonderlijk worden gekeken en de correcte gamma zal worden bepaald om de intensiteit op 128 te krijgen.
2.3.3 Totale normalisatie
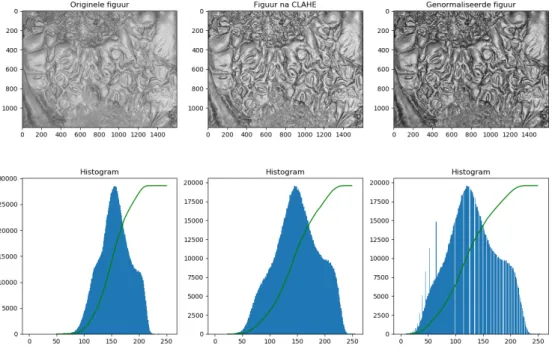
Nu alle normalisatie technieken zijn toegepast kan het uiteindelijke resultaat worden bekeken. In figuur 2.8 zal de originele figuur worden afgebeeld. Daarnaast zal de figuur worden afgebeeld na CLAHE en vervolgens wordt de uiteindelijk genormaliseerde figuur afgebeeld waarbij zowel CLAHE is toegepast als gamma transformatie.
16 HOOFDSTUK 2. DATA EXPLORATIE EN PREPROCESSING
2.4 Data augmentatie
Data normalisatie is belangrijk om elke afbeelding die in het model gaat zo constant mogelijk te krijgen. Dit zodat er geen over- of onderbelichte foto’s binnen gaan. En zodat het contrast bij elke binnen-gaande foto zo constant mogelijk is. Ondanks alle ondernomen pogingen om de input afbeeldingen zo constant mogelijk te maken zullen deze niet altijd constant zijn. Sommige afbeeldingen zullen nog steeds waziger, scherper, over- of onderbelicht zijn. Om het neuraal netwerk zo goed mogelijk met deze variaties te laten omgaan zal er aan data augmentatie worden gedaan. Data augmentatie zal de afbeeldingen aanpassen zodat deze niet meer genormaliseerd zijn. Dit kan gebeuren met dezelfde technieken als data normalisatie. Het histogram kan dus worden aangepast om de afbeelding terug wat over- of onderbelicht te maken. En ook de scherpte van de afbeelding kan worden aangepast met CLAHE. Een laatste techniek is de afbeeldingen wat uit te rekken. Zo zullen de huidmondjes veranderen van vorm. Er is gekozen om de augmentatie te doen op de genormaliseerde afbeeldingen en niet op de originele afbeeldingen. Deze keuze is gemaakt omdat het model uiteindelijk huidmondjes zal moeten herkennen op de genormaliseerde afbeeldingen en het model dus robust moet zijn op aanpassingen binnen de genormaliseerde afbeeldingen. Dit kan later nog worden aangepast wanneer blijkt dat het beter zou zijn om de augmentatie uit te voeren op de originele afbeelding.
2.4.1 Histogram equalization
De werking van histogram equalization is reeds onderzocht in sectie 2.3.1. In dat onderdeel werd er gebruik gemaakt van de CLAHE functie die de intensiteit van de pixelwaarde zal verdelen zodat alle intensiteiten evenveel gebruikt worden. Met de CLAHE functie zal het histogram ook worden uitgespreid om zo verder de intensiteiten te verdelen. In dit deel wordt er in eerste instantie gebruik gemaakt van een andere functie die ervoor zal zorgen dat het histogram meer of minder uitgespreid wordt. Er zal geen CLAHE meer worden uitgevoerd op de afbeelding omdat dit de geaugmenteerde data te ver zou verwijderen van de genormaliseerde data. Het is natuurlijk de bedoeling om een robuust model te bekomen en verdere augmentatie zou het model licht robuuster maken maar dit klein beetje extra robuustheid weegt niet op tegen de extra tijd dat het model nodig zou hebben voor de training. Te veel augmentatie zou de accuraatheid van het model zelfs naar beneden kunnen halen.
Het breder en smaller maken van het histogram zal gebeuren door elke pixel afzonderlijk met een waarde alfa te vermenigvuldigen. Deze waarde wordt random gekozen tussen 0,85 en 1,15. Wanneer de waarde groter is dan 1 dan zal het histogram breder worden en wanneer de waarde kleiner is dan 1 dan zal het histogram smaller worden. Deze functie zal ook de helderheid aanpassen. Dit is ongewenst dus zal de gamma tranformatie van sectie 2.3.2 worden toegepast om de helderheid weer te centreren.
2.4. DATA AUGMENTATIE 17
2.4.2 Gamma transformatie
Vervolgens zal de helderheid toch worden aangepast omdat dit een volgende vorm van data augmentatie is. Hier zal een random gamma worden gekozen tussen 0,75 en 1,25.
Nu zijn alle tranformaties op het histogram uitgevoerd en kan er worden gekeken naar transfor-maties op afmetingen binnen een figuur
2.4.3 Stretchen van de afbeelding
Een laatste augmentatie techniek is het stretchen van de afbeelding zelf. Het is namelijk mogelijk dat het gekozen blad iets rondere of ovalere huidmondjes heeft dan huidmondjes op andere bladeren van dezelfde soort. Om het model hierop te trainen zal de lengte van de afbeelding telkens met een random getal worden vermenigvuldigd. Dit getal zal zich tussen de 0,8 en 1,2 bevinden. Zo is het mogelijk om de afbeelding zowel in x als in y richting tot 20% uit te rekken. Dit zal altijd maar in één van de twee richtingen gebeuren omdat het niet nuttig is in beide richtingen tegelijk te stretchen.





![Figuur 3.7: Training loss (links) en test loss (rechts) op CIFAR-10 met 20-lagen en 56-lagen [4]](https://thumb-eu.123doks.com/thumbv2/5doknet/3292682.22061/51.892.146.723.301.498/figuur-training-loss-links-rechts-cifar-lagen-lagen.webp)
![Figuur 3.9: Het loss oppervlak van een ResNet-56 netwerk met en zonder skip connecties [5].](https://thumb-eu.123doks.com/thumbv2/5doknet/3292682.22061/52.892.132.779.166.437/figuur-loss-oppervlak-resnet-netwerk-skip-connecties.webp)